ആർക്കീയ
| ആർക്കീയ Temporal range: Paleoarchean – Recent
| |
|---|---|
| |
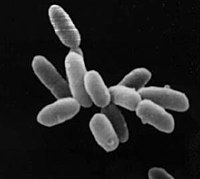
| ഹാലോബാക്റ്റീരിയ sp. strain NRC-1, ഓരോ കോശവും ഏകദേശം 5 μm നീളമുള്ളത്. | |
| ശാസ്ത്രീയ വർഗ്ഗീകരണം | |
| Domain: | ആർക്കീയ |
| ജന്തുലോകവും ഫൈലവും | |
|
റീനർക്കിയീട്ട | |
ഏകകോശ സൂക്ഷ്മജീവികളുടെ ഒരു കൂട്ടമാണ് ആർക്കീയ. (/ɑrˈkiːə/ ⓘ ar-KEE-ə ആർക്കീയോൺ എന്ന സാമ്രാജ്യത്തിലെ ഒരു ജീവി അല്ലെങ്കിൽ സ്പീഷീസുകളാണ് ഇവ. ഇതിനു കോശമർമ്മമോ കോശഭിത്തികളുള്ള അന്തർകോശവസ്തുക്കളോ ഇല്ല.
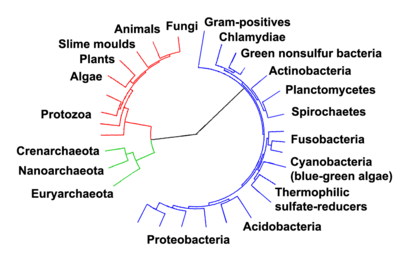
മൊണേറ കുടുംബത്തിലെ പ്രോക്കാരിയോറ്റസ്, ബാക്റ്റീരിയയായ അർക്കീബാക്റ്റീരിയായി പണ്ട് ഇവയെ കണക്കാക്കിയിരുന്നു, എന്നാൽ ഈ വർഗ്ഗീകരണം കാലഹരണപ്പെട്ടതായി[1] കണക്കാക്കുന്നു. ഇവയ്ക്ക് സ്വതന്ത്രമായ പരിണാമ മാറ്റങ്ങളും വഴികളുമുള്ളതും മറ്റു ജീവജാലങ്ങളുമായി ജൈവരാസഘടനയിൽ പലതരത്തിലുള്ള വ്യത്യാസവും കാരണം ഇപ്പോൾ ഇവയെ ത്രിതല സാമ്രാജ്യം പ്രത്യേകമായി വർഗ്ഗീകരിക്കുന്നു. ഈ ത്രിതലങ്ങളിൽ ശാരീരജൈവമാറ്റങ്ങൾ ഉൾക്കൊള്ളിച്ച് പരിണാമശാഖയെ ആർക്കീയ, ബാക്റ്റീരിയ, യൂക്കാരിയോട്ട എന്നിങ്ങനെ തരം തിരിക്കാം.
ആർക്കീയയെ തിരിച്ചറിഞ്ഞിട്ടുള്ള നാല് ഫൈലങ്ങളായി തരംതിരിക്കാം എന്നിരുന്നാലും വേറെയും ഫൈലങ്ങൾ ഉണ്ടാകാം. ഇവയിൽ ക്രെനാർക്കിയോട്ട, യൂരാർക്കിയോട്ട എന്നീ രണ്ടു വിഭാഗങ്ങളാണ് വിശദമായ പഠിക്കപ്പെട്ടത്. പ്രകൃതിയിൽ നിന്നും ലഭ്യമായ ഇവയുടെ മർമ്മാമ്ലം മൂലമാണ് ഇവയെ തിരിച്ചറിഞ്ഞിട്ടുള്ളത്, പരീക്ഷണ ശാലകളിൽ ഇതിനെക്കുറിച്ചുള്ള പഠനങ്ങൾ നടക്കാത്തതുകൊണ്ട് ഇവയുടെ വർഗ്ഗീകരണം ഇപ്പോഴും കഠിനമാണ്.
ആർക്കീയയും ബാക്റ്റീരിയയായും രൂപത്തിലും വലിപ്പത്തിലും വളരെ സാമ്യം പുലർത്തുന്നു, എന്നിരുന്നാലും ചിലത് അസാധാരണ ആകൃതി പുലർത്തുന്നു. ചിലത് ഹാലോക്വോഡ്രേറ്റം പോലെ ചതുരാകൃതിയുള്ളവയാണ്. ബാക്റ്റീരിയായോടുള്ള ഈ സാമ്യങ്ങൾക്കപ്പുറം, ജീനുകളും ഉപാപചയക്രമങ്ങളും യൂക്കാരിയോട്ടകളോട് സാമ്യമുള്ളതാണ്, പ്രത്യേകിച്ച് ന്യൂക്ലിക്കാസിഡ് ശൃംഖലകളോടും, ജീൻ പ്രോട്ടീൻ സിന്തസീസിനോടും, മാത്രമല്ല ഇതിലെ സമാന രാസാഗ്നികളും ഈ സാമ്യത്തിനുറപ്പേകുന്നു. ഇവയുടെ കോശസ്തരത്തിലെ ഈതർ ലിപ്പിഡുകൾ പോളുള്ളവയുടെ ജൈവരസതന്ത്രത്തിലെ പ്രത്യേകസ്വഭാവം ഇവയെ തികച്ചും വ്യത്യസ്തമാക്കുന്നു. ആർക്കീയ ഊർജ്ജം കണ്ടെത്തുന്നത് യൂക്കാരിയോട്ടുകളിൽ നിന്നും വ്യത്യസ്തമായ സ്രോതസ്സുകൾ ഉപയോഗിക്കുന്നു. ഓർഗാനിക്ക് സംയുക്തങ്ങളായ പഞ്ചസാര, അമോണിയ തുടങ്ങി ലോഹ അയോണുകളും ഇവ ഉപയോഗിക്കുന്നു, ഹൈഡ്രജൻ വാതകം പോലും ഊർജ്ജത്തിനായി ഉപയോഗിക്കുന്നു. ഉപ്പിനെ അതിജീവിക്കാൻ കഴിയുന്ന ആർക്കീയയായ ഹാലോഅർക്കിയ സൂര്യപ്രകാശത്തിനെ നേരിട്ട് ഊർജ്ജസ്രോതസ്സായി ഉപയോഗിക്കുന്നു, ചിലത് കാർബൺ ഫിക്സ് ചെയ്യുന്നു, എന്നാൽ സസ്യങ്ങളിൽനിന്നും സൈനോബാക്റ്റീരിയയിൽ നിന്നും വ്യത്യസ്തമായി അർക്കിയയിലെ ഒരു സ്പീഷീസും ഒരേ സമയം ഇതു രണ്ടും കൂടി ചെയ്യുന്നില്ല. ഇവ അലൈഗിക പ്രത്യുൽപ്പാദനം വഴി വംശവർദ്ധന നടത്തുന്നു, ദ്വിഖണ്ഡനം, ബഡ്ഡിംഗ്, ഫ്രാഗ്മെന്റേഷൻ, തുടങ്ങിയരീതികൾ അവലംബിക്കുന്നു. എന്നിരുന്നാൽ ബാക്റ്റീരിയ, യൂക്കാറ്റിയോട്ടകളിൽ നിന്നും വ്യത്യസ്തമായി, അർക്കിയയിലെ അറിയപ്പെടുന്ന ഒരു സ്പീഷീസും ബീജാണുക്കൾ ഉണ്ടാക്കുന്നില്ല.
തുടക്കത്തിൽ ഉപ്പു കായലുകളിലെ താപ നീരുറവകൾ പോലുള്ള പരുഷമായ ആവാസ വ്യവസ്ഥയിലാണ് ഇവ കാണപ്പെട്ടത്, പക്ഷെ പിന്നീടിവ മണ്ണ്, സമുദ്രം, നീർതടം, മനുഷ്യ ആവാസ വ്യവസ്ഥയിലും കാണപ്പെട്ടു. സമുദ്രങ്ങളിൽ ഇവ വളരെയധികം കാണപ്പെടുന്നു, പ്ലാങ്ക്ടണുകളിലെ അർക്കിയകൾ ഈ ഗ്രഹത്തിലെ തന്നെ സമ്പുഷ്ടമായ ജീവജാലങ്ങളിൽ ഒന്നാണ്. ഭൂമിയിലെ ഒരു പ്രധാന വിഭാഗമായി അർക്കിയ ഇപ്പോൾ കണ്ടുവരുന്നു. കാർബൺ നെട്രജൻ ചക്രങ്ങളിലും ഇവയ്ക്ക് പങ്കുണ്ടാകാം. പാരസൈറ്റുകളായോ പാതോജനുകളായോ ഇവയെ കാണുന്നതിനു വ്യക്തമായ തെളിവുകളില്ലെങ്കിലും മ്യൂട്ടലിസ്റ്റായും പരപോഷികളായും കാണുന്നു. ദഹേനേന്ദ്രിയ വ്യവസ്ഥയുടെ അവസാനത്ത് ഗുദഭാഗത്ത് കാണുന്ന മീഥനോജെനുകൾ ദഹനത്തിനു സഹായിക്കുന്നു. മീഥനോജെനുകൾ ജൈവ വാതകനിർമ്മാണത്തിനും അഴുക്കുചാലുകളുടെ ശുചീകരണത്തിനും മറ്റും ജൈവസാങ്കേതികവിദ്യ ഉപയോഗപ്പെടുത്തുന്നു.
വർഗ്ഗീകരണം[തിരുത്തുക]
പുതിയ സാമ്രാജ്യം[തിരുത്തുക]

ജൈവരസതന്ത്രം, ആകൃതി, ഉപാപചയം തുടങ്ങിയവയുടെ അടിസ്ഥാനത്തിൽ 20-ആം നൂറ്റാണ്ടിൽ, പ്രോക്കാരിയോട്ടകൾ ഒരേ കൂട്ടം ജീവികളായി വർഗ്ഗീകരിച്ചിരുന്നത്. ഉദാഹരണത്തിന്, സൂക്ഷ്മാണു ശാസ്ത്രജ്ഞന്മാർ ശരീരത്തിന്റെ ആകൃതി, അവയുടെ കോശഭിത്തിയുടെ ആകൃതി, കഴിക്കുന്ന ആഹാരം തുടങ്ങിയവയെ അടിസ്ഥാനപ്പെടുത്തിയാണ് വർഗ്ഗീകരണം നടത്തിയത്.[2] എന്നാൽ 1965[3]-ൽ ശാസ്ത്രജ്ഞന്മാർ പുതിയ ഒരു സമീപനം കൊണ്ടുവന്നു, ജീനുകളുടെ നിരകൾ അടിസ്ഥാനമാക്കി ഏതേതു ജീവികൾ സാധുതയോടുകൂടി പരസ്പരം ബന്ധപ്പെട്ടിരിക്കുന്നു എന്നു കണ്ടെത്തി. ഈ സമീപനത്തെ ഫൈലോജനിറ്റിക്സ് എന്നു പറയുന്നു, ഇതേ രീതി പിൽക്കാലങ്ങളിൽ തുടർന്നുപോന്നു.
അർക്കിയ വംശവൃക്ഷത്തിൽ പ്രോക്കാരിയോട്ടെയിലെ പുതിയ വിഭാഗമായി വർഗ്ഗീകരിച്ചത് 1977 കാൾ വോസീ, ജോർജ്ജ് ഇ. ഫോക്സ് എന്നിവർ റൈബോസോമൽ ആർ.എൻ.എ (rRNA) ജീനുകളുടെ[4] അനുക്രമത്തിനെ അടിസ്ഥാനമാക്കിയാണ്. ഈ രണ്ടു വിഭാഗങ്ങൾ നാമകരണം ചെയ്തത് അർക്കിബാക്റ്റീരിയ, യൂബാക്റ്റീരിയ എന്നായിരുന്നു, ജന്തുലോകം ഉപ ജന്തുലോകം എന്നിവയായി വോസിയും ഫോക്സും പരിഗണിച്ചത്. വോസി ഇവ പുതിയ തരം ജീവി വിഭാഗമാണെന്നു വാദിച്ചു. ഈ മാറ്റങ്ങൾക്ക് ഊന്നൽ നൽകാൻ ഈ വിഭാഗങ്ങളെ പിന്നീട് അർക്കിയ എന്നും ബാക്റ്റീരിയ എന്നും പുനർനാമകരണം ചെയ്തു[5]. അർക്കിയ എന്ന പേര് പുരാതന ഗ്രീക്ക് പദവും പുരാതന വസ്തു എന്ന് അർത്ഥം വരുന്ന ἀρχαῖα ൽ നിന്നാണ്. [6]
തുടക്കത്തിൽ മീഥനോജെനുകൾ മാത്രമാണ് ഈ പുതിയ സാമ്രാജ്യത്തിൽ ഉൾപ്പെടുത്തിയത്, അർക്കിയകൾ ഉപ്പു കായലുകളിലെ താപ നീരുറവകൾ പോലുള്ള പരുഷമായ ആവാസ വ്യവസ്ഥയിലാണ് ഇവ കാണപ്പെട്ടത്. 20-ആം നൂറ്റാണ്ടിന്റെ അവസാനത്തോടെ പ്രകൃതിതിയിലെമ്പാടും പടർന്നുകിടക്കുന്നവയാണിതെന്ന് കണ്ടെത്തുകയും, പിന്നീടിവ മണ്ണ്, സമുദ്രം, നീർതടം എന്നിവിടങ്ങളിലും കാണപ്പെടുന്നതായും കണ്ടെത്തി[7]. പക്ഷെ ഈ കണ്ടെത്തൽ പ്രകൃതിയിൽ നിന്നും ലഭ്യമായ ഇവയുടെ മർമ്മാമ്ലം മൂലമാണ്, പരീക്ഷണ ശാലകളിൽ ഇതിനെ വളർത്തിയെടുത്തിട്ടില്ല.[8][9]
ഇപ്പോഴുള്ള വർഗ്ഗീകരണം[തിരുത്തുക]

തുടർച്ചയായി പെട്ടെന്ന് മാറിക്കൊണ്ടിരിക്കുന്നതാണ് അർക്കിയയുടേയും പ്രോക്കാരിയോട്ടകളുടെയും വർഗ്ഗീകരണം. ഇപ്പോഴുള്ള വർഗ്ഗീകരണം ലക്ഷ്യം വെച്ചിരിക്കുന്നത് ഇവയുടെ പൂർവ്വികരുടെ ആകൃതിയിൽ സ്വഭാവഗുണങ്ങൾ ഒത്തുനോക്കി കൂട്ടമാക്കാനാണ്.[10] ഇതു റൈബോസോമൽ ആർ.എൻ.എ (rRNA) ജീനുകളുടെ അനുക്രമം ചേർത്തുവെച്ച് ജീവികൾ തമ്മിലുള്ള സാദൃശ്യം നോക്കിയാണ്.[11] പരീക്ഷണശാലയിൽ വളർത്താവുന്നതും ശരിയായി പഠനത്തിനു വിധേയമായതുമായ അർക്കിയകൾ രണ്ട് പ്രധാന ഫൈലങ്ങളായ യൂര്യാർക്കിയോട്ട, ക്രേനാർക്കിയോട്ട എന്നിവയിൽ പെടുത്തിയിരിക്കുന്നു. മറ്റുള്ളവയെ പ്രഥമയത്നാത്മകമായി ഉണ്ടാക്കിയവയാണ്, ഉദാഹരണമായി, 2003 ൽ കണ്ടെത്തിയ നാനോർക്കിയോം ഇക്വിറ്റൻ അതിനായി തന്നെ ഒരു ഫൈലം കൊടുത്തിരിക്കുന്നു.[12] ഒരു പുതിയ ഫൈലമായ കൊരാർക്കിയോട്ട നിർദ്ദേശിച്ചിട്ടുമുണ്ട്. ഈ ഫൈലം അസാമാന്യമായ ഒരു കൂട്ടം അതീവതാപശേഷിയുള്ള സ്പീഷീസുകൾ ആണ്, ഇവയ്ക്ക് മാതൃ ഫൈലത്തിനോട് സാദൃശ്യങ്ങളും എന്നാൽ ക്രേനാർക്കിയോട്ടകളോട് അതീവ സാദൃശ്യവുമുണ്ട്.[13][14] അടുത്തിടെ കണ്ടെത്തിയ മറ്റുള്ള സ്പീഷീസുകൾ വളരെ വിദൂരസാദൃശ്യം പ്രകടിപ്പിക്കുന്നതുമാണ്, ഉദാഹരണത്തിന് 2006-ൽ[15] കണ്ടെത്തിയ അർക്കിയൽ റിച്ച്മണ്ട് മൈൻ അസിഡോഫിലിക് (ARMAN) പോലുള്ളവ. ഇവ ലോകത്തിലെ ഏറ്റവും ചെറിയ ജീവജാലങ്ങളിലൊന്നാണ്.[16]
സ്പീഷീസ്[തിരുത്തുക]
സ്പീഷീസുകളുടെ വർഗ്ഗീകരണവും തർക്കവിഷയമാണ്. ജീവശാസ്ത്രം നിർവ്വചിച്ചിരിക്കുന്നത് ഒരു കൂട്ടം സദൃശ ജീവജാലത്തിന്റെ കൂട്ടമാണ് സ്പീഷീസ് എന്നാണ്. പ്രത്യുല്പാദനം സ്വവർഗ്ഗത്തിനോടുമാത്രം എന്നുള്ളത് അർക്കിയകളിൽ ബാധമല്ലാത്തതിനാൽ (ഇവ അലൈഗിക പ്രത്യുൽപ്പാദനം നടത്തുന്നു[17]) ഇത് യോജിക്കുന്നില്ല.
അർക്കിയ തിരശ്ചീന ജീൻ കൈമാറ്റം വളരെ തീവ്രമായ രീതിയിൽ നടത്തുന്നു. ചില ശാസ്ത്രജ്ഞന്മാർ സൂചിപ്പിക്കുന്നതു, ജനുസ് ഫെറൊപ്ലാസ്മയിൽ കാണുന്നതുപോലെ ജനിതകത്തിൽ അടുത്ത ബന്ധം പുലർത്താത്ത തമ്മിൽ ജീൻ കൈമാറ്റം നടത്തി ഉണ്ടാകുന്ന സ്പീഷീസുകളെ കൂട്ടമാക്കാം.[18]. ഹലൊറുബ്രത്തിൽ നടത്തിയ പഠനം അടുത്ത ബന്ധം പുലർത്താത്തവ തമ്മിൽ ജീൻ കൈമാറ്റം നടത്തിയതായ കാണുന്നു, ഇത് വർഗ്ഗീകരണത്തിന്റെ സാധുത ചെറുതാക്കുന്നു.[19] എന്നാൽ ഇവയുടെ വർഗ്ഗീകരണത്തിന്റെ പ്രാവർത്തിക പ്രാധാന്യമെത്രത്തോളമെന്ന് ചിന്തിക്കേണ്ടിയിരിക്കുന്നു.[20]
അർക്കിയ ജനിതക വൈചിത്ര്യം കാരണം വ്യക്തമായ രീതിയിൽ സ്പീഷിസുകൾ എത്രയെന്നു പറയുവാൻ കഴിയില്ല.[11] കണക്കുകൾ സൂചിപ്പിക്കുന്നത് ഫൈലങ്ങൾ 18 മുതൽ 23 വരെയുണ്ടാകാം, ഇതിൽ 8 എണ്ണം മാത്രമേ പരീക്ഷണ ശാലകളിൽ പുനഃ നിർമ്മിക്കുകയും നേരിട്ട് പഠന വിഥേയമാക്കുകയും ചെയ്തിട്ടുള്ളൂ. ഇവയിൽ മിക്കവയും ഒരു rRNA അനുക്രമത്തിൽ നിന്നും ഊഹിച്ചെടുത്തതിനാൽ, ഇവയിലെ വൈചിത്ര്യത്തിനുള്ള സാധ്യത മങ്ങുന്നു.[21] ബാക്റ്റീരിയയ്ക്കും കൾച്ചർചെയ്യാൻ കഴിയാത്ത അനേകം സാമ്യമുള്ള ജനിതകങ്ങൾ ഉൾക്കൊള്ളുന്ന മൈക്രോബുകൾ ഉള്ളതും സ്വഭാവനിർണ്ണയം വഴിമുട്ടുന്നു.[22]
ആവിർഭാവവും പരിണാമവും[തിരുത്തുക]
പ്രോക്കാരിയോട്ടകളുടെ കോശ ജീവാശ്മം 350 കോടി വർഷങ്ങൾ മുൻപുതന്നെ കണ്ടെത്തിയെങ്കിലും മിക്കവയുടെയും ആകൃതി അർക്കിയകളെ കണ്ടെത്തുന്നതിനുപയോഗിക്കാൻ സാധിക്കില്ല.[23] എന്നാൽ രാസീയ ഫോസിലുകളിലെ ലിപ്പിടുകൾ അധിക വിവരങ്ങൾ നൽകുന്നു, ഇവ മറ്റ് ജീവികളുടെ ഫോസിലുകളിൽ കാണുന്നതല്ല.[24] 270 കോടി വർഷങ്ങൾക്കു മുൻപ് അർക്കിയകളുടെയും യൂക്കാരിയോട്ടകളുടെയും ശിഷ്ടങ്ങൾ പാറകളിൽ കണ്ടെത്തിയതായി ചില പ്രസിദ്ധീകരണങ്ങൾ അവകാശപ്പെട്ടിരുന്നു;[25] എന്നാൽ ഈ അവകാശവാദങ്ങൾ ചോദ്യംചെയ്യപ്പെടുകയും ചെയ്തു.[26] പ്രികാബ്രിയൻ രൂപീകരണത്തിൽ ഇതുപോളുള്ള ലിപ്പിഡുകളെ കണ്ടെത്തിയിട്ടുണ്ട്. 380 കോടി വർഷങ്ങൾക്ക് മുൻപ് ഉണ്ടായതും ഏറ്റവും പഴക്കം ചെന്ന എക്കൽ ശേഖരമായ വെസ്റ്റ് ഗ്രീൻലാൻഡിലെ ഇസുവ ജില്ലയിൽ ഇത്തരത്തിലെ ഏറ്റവും പഴക്കം ചെന്ന ലിപ്പിഡിനെ കണ്ടെത്തിയിട്ടുണ്ട്.[27] ഭൂമിയിലെ ഏറ്റവും പഴക്കം ചെന്ന ജീവവംശം അർക്കിയ ആയിരിക്കാം.[28]
വോസീ അവകാശപ്പെടുന്നതെന്തെന്നാൽ ബാക്റ്റീരിയ, അർക്കിയ, യൂക്കാരിയോട്ടസ് തുടങ്ങിയവ ഒരു ജീവജാല കോളനിയുടെ പരിണാമ വഴികൾ, പുരാതനകാലത്ത് വ്യത്യസ്തദിശകളിൽ തിരിഞ്ഞതാണെന്നാണ്.[30][31] എന്നിരുന്നാലും ചില ശാസ്ത്രജ്ഞർ വാദിക്കുന്നത് അർക്കിയയും യൂക്കാരിയോട്ടകളും ബാക്റ്റീരിയയിൽ നിന്നും ഉരുത്തിരിഞ്ഞതാണെന്നാണ്.[32] എങ്ങനെയായാലും, വൈറസുകളും അർക്കിയകളും ഏകദേശം 200 കോടി വർഷങ്ങൾ മുൻപാണ് ബന്ധപ്പെടാൻ തുടങ്ങിയത്, ഇവയുടെ പരിണാമം ഈ കൂട്ടത്തിലെ അംഗങ്ങളിൽനിന്നു തന്നെയാകും.[33] ബാക്റ്റീരികളും അർക്കിയകളും പൂർവ്വികർ തെർമോഫൈലുകളാകാൻ സാധ്യതയുണ്ട്, ഇവയ്ക്ക് താഴ്ന്ന താപനിലകളാകാം അസഹനീയം, താഴ്ന്ന താപനിലകളിൽ വളരുന്ന ജീവജാലങ്ങൾ പിന്നീട് വന്നതുമാകാം.[34] അർക്കിയയും ബാക്റ്റീരിയയും തമ്മിൽ ചേരാത്തതിനാലും, പ്രൊക്കാരിയോട്ടെന്ന പദം യൂക്കാരിയോട്ടല്ല എന്ന അർഥത്തിൽ മാത്രമേ കാണാൻ സാധിക്കുള്ളൂ.[35]
യൂക്കാറ്റിയോട്ടസിനോടുള്ള ബന്ധം[തിരുത്തുക]
അർക്കിയക്ക് യൂക്കാറ്റിയോട്ടകളോടുള്ള പരിണാമത്തിലെ സമാനതകൾ വ്യക്തമല്ല. മറിച്ച് കോശാകൃതി, പ്രവർത്തനഗുണങ്ങളും താഴെപ്പറയുന്ന പല ജനിതക വൃക്ഷങ്ങളും ഇവയെ ഒരേ കൂട്ടമായി കാണിക്കുന്നു.
യൂരിയാർക്കിയോട്ടയും ഫൈലം ക്രേനർക്കിയോട്ടകളും തമ്മിലുള്ള ചേർച്ചയേക്കാൾ ആർക്കിയയുടെ ഫൈലം ക്രേനർക്കിയോട്ടകളും യൂക്കാരിയോട്ടകളും തമ്മിൽ ചേർച്ച കാണുന്നു,[36] മാത്രമല്ല ജീൻ കൈമാറ്റം മൂലം അർക്കിയയുടെ ജീനുകൾ പോലുള്ളവ തെർമറ്റോഗാ മരിറ്റിമയെ പോലുള്ള ബാക്റ്റീരിയകളിൽ കാണപ്പെടുന്നു. ഇപ്പോൾ മുന്നിൽ നിൽക്കുന്ന ദൃഷ്ടികോണുകൾ സൂചിപ്പിക്കുന്നത് യൂക്കാറ്റിയോട്ടസ് ഉരുത്തിരിഞ്ഞത് അർക്കിയകളിൽ നിന്നാണെന്നാണ്[37][38], ഈ യൂക്കാരിയോട്ടകൾ ഉണ്ടായത് അർക്കിയയും യൂബാക്റ്റീരിയവും തമ്മിലുള്ള കൂടിച്ചേരലിന്റെ ഫലമായാണ്, ഇത് കോശമർമ്മവും കോശാന്തർഭാഗവുമായി; പക്ഷെ ഈ വാദത്തിന് ജെനിതകത്തിനെ വിശദീകരിക്കുവാൻ കഴിയുമെങ്കിലും, കോശഘടന വിശദീകരിക്കാൻ ബുദ്ധിമുട്ടുണ്ടാക്കുന്നു.[39]
ഘടന[തിരുത്തുക]

ഒറ്റയായ അർക്കിയ 0.1 മൈക്രോമീറ്റർ (μm) മുതൽ 15 μm വരെ വ്യാസമുള്ളവയാണ്, ഗോളം, ദണ്ഡ്, അണ്ഡാകൃതി അല്ലെങ്കിൽ പരന്നതോ ആയി പലരൂപത്തിൽ ഇവയെ കാണുന്നു.[40] മറ്റു ചില ആകൃതികൾ ക്രേനർക്കിയോട്ടകൾ വ്യത്യസ്തവും ക്രമരഹിതവും സൾഫൊലോബസ് പോലെ ദളോപമമായും, തെർമോഫിലം പോലെ അര മൈക്രോണിനു താഴെ വ്യാസം വരുന്ന സൂചിപോലുള്ള ഫിലമെന്റുകൾ, തെർമോപ്രോട്ടെസ്, പൈറോബാക്കുലം പോലെ പൂർണ്ണ ചതുരാകൃതിയിലും കാണുന്നു.[41] അധികമായ ഉപ്പുവെള്ളത്തിൽ കഴിയുന്ന സമചതുരാകൃതിയുള്ള അർക്കിയ ഹാലോക്വോഡ്രേറ്റം വാൽസിബി പരന്നതാണ്.[42] ഈ വ്യത്യസ്തമായ ആകൃതി ഇവയുടെ കോശഭിത്തിയും പ്രോക്കാരിയോട്ടിക്ക് സൈറ്റോസ്കെൽറ്റോൺ ഉള്ളതിനാലാണ്. മറ്റ് ജീവജാലങ്ങളുടെ പ്രോട്ടീൻ സംബന്ധമായ സൈറ്റോസ്കെൽറ്റോൺ ഘടകഭാഗം അർക്കിയയിലുണ്ട്,[43] കോശത്തിനുള്ളിൽ നിന്നും ഫിലമെന്റുകളുമുണ്ട്,[44] പക്ഷെ മറ്റു ജീവികളെ അപേക്ഷിച്ച് ഇവ എന്താണെന്ന് മനസ്സിലാക്കിയിട്ടില്ല.[45] തെർമൊപ്ലാസ്, ഫെറോപ്ലാസ്മ എന്നിവയിലെ കോശഭിത്തികളില്ലാത്തതിനാൽ ക്രമരഹിതമായ ആകൃതി കാണുന്നു, ഇവയ്ക്ക് അമീബിയ അനുരൂപമുണ്ട്.[46]
ചില സ്പീഷീസുകളിൽ ഫിലമെന്റുകളോ മൊത്തമായോ കോശങ്ങൾ 200 μm വരെ നീളമുള്ളതാണ്.[40] ബയോഫിലിൽ ഔന്നത്യമുള്ളവയാണീ ജീവികൾ.[47] പ്രത്യേകിച്ച് തെർമ്മോകോക്കസ് കൊലിസെൻസ് കോശങ്ങൾ പറ്റിച്ചേർന്ന് ഒരു വലിയ കോശമായിത്തീരുന്നു.[48] അർക്കിയ ജനുസ്സ് പൈറോഡിക്റ്റിയം പോലുള്ളവയിൽ കുഴൽ പോലുള്ളതും കോശങ്ങൾക്കു പുറത്തേക്കു നിൽക്കുന്ന കനം കുറഞ്ഞതും നീളമുള്ള കന്യൂലെ കോശങ്ങളുടെ കോളനിയായി അഗലൊമെരിയേഷൻ പോലുള്ള പൊന്തച്ചെടി കാണുന്നു.[49] ഇവയുടെ ഉപയോഗം വ്യക്തമല്ലെങ്കിലും, അടുത്തുള്ളവയുമായി ആശയവിനിമയം നടുത്തുവാനും പോഷകങ്ങൾ പങ്കുവെയ്ക്കാനോ ഉപയോഗിക്കുന്നു.[50] 2001 ൽ ജർമ്മൻ സ്വാമ്പിൽ കണ്ടെത്തിയ കൂട്ടത്തിൽ വിവിധ സ്പീഷിസുകൾ കാണപ്പെട്ടു, സ്പ്രിങ്ങ് ഓഫ് പേൾസ് പോലുള്ളവയാണിത്. ഉരുണ്ടതും വെളുത്തതും യൂക്കാരിയോട്ട സ്പീഷീസ് ഫിലമെന്റുകളായി നീളത്തിൽ 15 സെ.മീ വളരുന്നു; ഈ ഫിലമെന്റുകൾ ചില പ്രത്യേക ബാക്റ്റീരിയ സ്പീഷിസിനാൽ നിർമ്മിതമാണ്.[51]
ആകൃതിയും പുരോഗതിയുടെ സംഗ്രഥനവും പ്രവർത്തനവും[തിരുത്തുക]
അർക്കിയകൾക്കും ബാക്റ്റീരിയകൾക്കും ഏകദേശം ഒരുപോലുള്ള കോശഘടനയാണുള്ളത്, എന്നാൽ കോശ നിർമ്മാണഘടകങ്ങൾ അർക്കിയയെ വ്യത്യസ്തമാക്കുന്നു. ബാക്റ്റീരിയയെപ്പോലെ അന്തർകോശസ്തരവും അന്തർകോശവസ്തുക്കളോ ഇല്ല.[35] കോശഭിത്തിയാൽ ചുറ്റപ്പെട്ട ഇവ ഒന്നോ അതിലധികമോ ഫ്ലാഗല്ലകൾ ഉപയോഗിച്ച് നീന്തുന്നു.[52] ഘടനാപരമായി ഗ്രാം പോസിറ്റീവ് ബാക്റ്റീരിയയുമായി വളരെയധികം സാമ്യം പുലർത്തുന്നു. മിക്കവയ്ക്കും ഒരു പ്ലാസ്മാ മെബ്രേനും, കോശഭിത്തിയുമേ ഉള്ളൂ, കോശസ്തര വിടവ് ഇവയിൽ കാണപ്പെടുന്നില്ല. ഈ പൊതുനിയമങ്ങൾ മാറ്റിവെച്ച് ഇഗ്നികോക്കസിന് വലിയ പെരിപ്ലാസവും സ്തരത്താൽ പൊതിഞ്ഞ വാസിക്കിളുകൾ തൊലിയാൽ മൂടപ്പെട്ടും കാണുന്നു.[53]
സ്തരം[തിരുത്തുക]

മറ്റ് ജീവികളിൽ നിന്നും തികച്ചും വ്യത്യസ്തമായ തന്മാത്രകളാൽ നിർമ്മിതമായ സ്തരമാണ് അർക്കിയകളുടേത്, ഇവ ബാക്റ്റീരിയയിൽ നിന്നും യൂക്കാരിയോട്ടകളിൽ നിന്നും വിദൂരസാദൃശ്യം മാത്രമേ ഉള്ളൂ എന്നാണ് ഇതു കാണിക്കുന്നത്.[54] എല്ലാ ജീവികളിലും കോശസ്തരം നിർമ്മിച്ചിരിക്കുന്നത് ഫോസ്ഫോലിപ്പിഡ് തന്മാത്രകളാലാണ്. ഈ തന്മാത്രകളുടെ പോളാർ ഭാഗങ്ങളിൽ (ഫോസ്ഫേറ്റ് തല ഭാഗം) ഒരു ഭാഗം ജലത്തിൽ ലയിക്കുന്നതും, നോൺ പോളാർ (ലിപ്പിഡ് വാല്) ഭാഗം ലയിക്കാത്തതുമാണ്. ഈ രണ്ടുഭാഗങ്ങൾ ഗ്ലിസറോൾ കൊണ്ട് ബന്ധിപ്പിച്ചിരിക്കുന്നു. ജലത്തിൽ, ഫോസ്ഫോലിപ്പിഡുകളുടെ കുലകൾ തല ജലത്തിലേക്കും വാല് അതിനെതിരായും കാണുന്നു. ലിപ്പിഡ് ബൈലെയർ എന്നറിയപ്പെടുന്ന ഫോസ്ഫോലിപ്പിഡുകളുടെ രണ്ടു പാളികൾ ചേർന്ന് നിർമ്മിക്കപ്പെട്ടിരിക്കുന്നതാണ് കോശത്തിലെ പ്രധാന ആകൃതി.
ഈ ഫോസ്ഫോലിപ്പിഡുകൾ നാലു രീതിയിൽ വൈചിത്ര്യം കാണിക്കുന്നു:
- ബാക്റ്റീരിയയുടേയും യൂക്കാരിയോട്ടകളുടേയും സ്തരം രൂപീകൃതമായിരിക്കുന്നത് മുഖ്യമായും ഗ്ലൈക്കോൾ-എസ്റ്റർ ലിപ്പിഡുകൾ കൊണ്ടാണ്, എന്നാൽ അർക്കിയകളുടേത് ഗ്ലൈക്കോൾ-ഈഥർ ലിപ്പിഡുകൾ കൊണ്ടാണ്.[55] തമ്മിലുള്ള വ്യത്യാസം ലിപ്പിഡുകൾ ഗ്ലിസറോൾ സമാംശവും തമ്മിൽ ബന്ധിപ്പിക്കുന്ന ബന്ധനം ആണ്. രണ്ടു ബന്ധനങ്ങളും വലതുവശത്തെ ചിത്രത്തിൽ മഞ്ഞ നിറത്തിൽ കാണിച്ചിരിക്കുന്നു. എസ്റ്റർ ലിപ്പിഡിൽ എസ്റ്റർ ബന്ധനവും, ഈഥർ ലിപ്പിഡിൽ ഈഥർ ബന്ധനവുമാണ്. എസ്റ്റർ ബന്ധനത്തേക്കാൾ ഈഥർ ബന്ധനം രാസപരമായി ശക്തമാണ്. ഈ ഗുണം അർക്കിയകളെ വളരെ ഉയർന്ന ഊഷ്മാവിലും, ഉയർന്ന അമ്ലാന്തരീക്ഷത്തിലോ, ക്ഷാരാന്തരീക്ഷത്തിലോ നിലനിൽക്കാൻ സഹായിക്കുന്നത്.[56] ബാക്റ്റീരിയയിലും യൂക്കാരിയോട്ടകളും ചില ഈഥർ ലിപ്പിടുകൾ കാണുന്നു, പക്ഷെ അർക്കിയകളിലെ സ്തരത്തിൽ ധാരാളമായി കാണപ്പെടുന്നില്ല.
- ഗ്ലിസറോൾ സമാംശത്തിന്റെ സ്റ്റീരിയോ കെമസ്ട്രി മറ്റുള്ള ജീവിയുടേതിൽ നിന്നും വിപരീതമാണ്. ഗ്ലിസറോൾ സമാംശം രണ്ട് രീതിയിൽ ഉണ്ടാകാം ഒന്നിന്റെ പ്രതിബിംബം പോലെ, ഇവയെ റൈറ്റ്ഹാർഡഡ്, ലെഫ്റ്റ്ഹാർഡഡ് ഫോംസ്(right-handed and left-handed forms) എന്നു വിളിക്കുന്നു. രസതന്ത്രത്തിൽ ഇവയെ എനാന്റിയോമേർസ് എന്നു പറയുന്നു. ബാക്റ്റീരിയയും യൂക്കാരിയോട്ടുകളിൽ നിന്നും വ്യത്യസ്തമായി ഈ രണ്ടും വ്യത്യസ്ത സ്വഭാവവും ഉണ്ടാക്കുന്ന രാസാഗ്നിയും വ്യത്യസ്തമാണ്. ഇത് സൂചിപ്പിക്കുന്നതെന്തെന്നാൽ ബാക്റ്റീരിയയും യൂക്കാരിയോട്ടുകളും ഫോസ്ഫോലിപ്പിഡുകൾ ഉണ്ടാക്കാൻ ഉപയോഗിക്കുന്ന രാസാഗ്നിയിൽ നിന്നും തികച്ചും വ്യത്യസ്തമായാണ് അർക്കിയ ഉപയോഗിക്കുന്നത്. ഈ തരത്തിലെ രാസാഗ്നികൾ വളരെ മുൻപുതന്നെ പരിണാമവഴിയിൽ ഉണ്ടാക്കിയിട്ടുണ്ട്, അതിനാൽ ഈ മുൻപുതന്നെ ഈ രണ്ടു സാമ്രാജ്യങ്ങൾ വേർപിരിഞ്ഞതാണെന്നു കാണാം.[54]
- അർക്കിയയിലെ ലിപ്പിഡുകൾ മറ്റു ജീവികളിൽ നിന്നും രാസപരമായി വ്യത്യാസപ്പെട്ടിരിക്കുന്നു. വശങ്ങളിൽ ധാരാളം ശാഖകളോടുകൂടിയതും നീളമുള്ള ചങ്ങലയോടുകൂടിയതുമായ ഐസോപ്രിനോയിഡ് ശാഖകളുള്ളവയാണിത്, ചിലപ്പോൾ സൈക്ലോപ്രൊപ്പൈൻ, സൈക്ലോഹെക്സൈൻ വലയങ്ങളോടുകൂടിയതുമാണ്.[57] മറ്റു ജീവജാലങ്ങളിലെ സതരങ്ങളിൽ ഫാറ്റി ആസിഡുകൾക്ക് ശാഖകളോ വലയങ്ങളോ ഇല്ലാത്ത ഒരേ ഒരു നേരേയുള്ള ചങ്ങലയാണുള്ളത്. ഐസോപ്രിനോയിഡ് പല ജീവികളുടെയും ജൈവരസതന്ത്രത്തിൽ ഒരു പ്രധാന പങ്കുവഹിക്കുന്നു, അർക്കിയ മാത്രമാണ് ഫോസ്ഫോലിപ്പിഡുകളുടെ നിർമ്മാണത്തിനിതുപയോഗിക്കുന്നത്. ഈ ശാഖാ ചങ്ങലയാകാം ഇവയെ ഉയർന്ന ഊഷ്മാവിൽ നിലനിൽക്കാൻ സഹായിക്കുന്നത്.[58]
- ചില അർക്കിയകളിൽ ലിപ്പിഡ് ബൈലെയറുകൾക്ക് പകരം മോണോലെയർ. ഫലമായി, രണ്ടു അർക്കിയ ഫോസ്ഫോലിപ്പിഡ് വാലുകൾ ചേർന്ന് ഒരു തന്മാത്രയാകുകയും ഇവയ്ക്ക് രണ്ട് തലകൾ ആകുകയും ചെയ്യുന്നു. ഇതിനാൽ സ്തരം ശക്തിയുള്ളതാകുകയും പരുഷമായ സാഹചര്യങ്ങളിൽ നിലനിൽക്കാൻ സഹായിക്കുന്നതും.[59] ഉദാഹരണത്തിന്, ഫെറോപ്ലാസ്മ ഈ തരത്തിൽപെടുന്നവയാണ്, ഇവയ്ക്ക് ഉയർന്ന അമ്ലാവസ്ഥയിലെ ആവാസവ്യവസ്ഥയിൽ നിലനിൽക്കാൻ കഴിയുന്നു.[60]
ഭിത്തിയും ഫ്ലജല്ലയും[തിരുത്തുക]
മിക്ക അർക്കിയകളിലും (തെർമോപ്ലാസ്മയും ഫെറോപ്ലാസ്മയുമൊഴിച്ച്) കോശഭിത്തി ഉണ്ട്.[46] മിക്ക അർക്കിയകളിലും ഉപരിതല പ്രോട്ടീൻ പാളി കോശഭിത്തിയിലുണ്ട്, ഇതൊരു S-പാളി ഉണ്ടാക്കുന്നു.[61] S-പാളി പ്രോട്ടീനുകൾ കൊണ്ട് കോശത്തെ പൊതിഞ്ഞ കട്ടികൂടിയ ആവരണമാണ്.[62] ഈ ആവരണപാളി രാസ ഭൗതിക സംരക്ഷണം നൽകുന്നു, മറ്റു സൂക്ഷ്മതന്മാത്രകൾ കോശ സ്തരവുമായി സമ്പർക്കത്തിൽ വരുന്നതു തടയുന്നു.[63] ബാക്റ്റീരിയയെപ്പോലെയല്ലാതെ അർക്കിയയിലെ കോശഭിത്തിയിൽ പെപ്റ്റിഡൊഗ്ലൈക്കാൻ ഇല്ല.[64] മെഥനോബാക്റ്റീരിയകൾക്ക് സ്യൂഡോപെപ്റ്റിഡോഗ്ലൈക്കാൻ ഉള്ള കോശഭിത്തിയുണ്ട്, രാസഘടനയിലുള്ള വ്യത്യാസമൊഴിച്ച്, പ്രവർത്തനത്തിലും, ഭൗതിക ഘടനയിലും യൂബാക്റ്റീരിയൽ പെപ്റ്റിഡോഗ്ലൈക്കാനോട് സമാനതയുണ്ട്, ഇവയിൽ D-അമിനോ ആസിഡ്, N-അസറ്റൈൽമ്യുറാമിക്ക് ആസിഡ് എന്നിവ കാണുന്നില്ല.[63]
അർക്കിയയിലെ ഫ്ലജെല്ല പ്രവർത്തിക്കുന്നത് ബാക്റ്റീരിയയിലെ പോലെയാണ്, ഇവയുടെ നീളമുള്ള തണ്ട് മൂലത്തിൽനിന്നും റോട്ടറി മോട്ടോറുകളാൽ പ്രവർത്തിക്കുന്നു. സ്തരത്തിലങ്ങളോമുള്ള പ്രോട്ടോൺ വിന്യാസമാണ് ഇതിനുള്ള ഊർജ്ജം പ്രദാനം ചെയ്യുന്നത്. പക്ഷെ പുരോഗതിയും രീതിയിലും ഇവ വ്യത്യസ്തമാണ്.[52] രണ്ടു തരത്തിലെയും ഫ്ലജല്ലകളും വ്യത്യസ്തമായ പൂർവ്വികരിൽനിന്നും ഉണ്ടായവയാണ്. ബാക്റ്റീരിയകളുടെ ഫ്ലജല്ലം റ്റയിപ്പ് III സെക്രീഷൻ സിസ്റ്റം ഉള്ള ഒരു പൊതു പൂർവ്വികരെ പങ്കുവെയ്ക്കുന്നു,[65][66] എന്നാൽ അർക്കിയയുടെ ഫ്ലജല്ലം ഉണ്ടായത് ടൈപ്പ് IV പെലസിൽ നിന്നാണ് ഉണ്ടായതാണ്.[67] ബാക്റ്റീരിയയുടെ ഫ്ലജല്ലം പൊള്ളയായതും ഉപശാഖ അറ്റത്തേക്കു സഞ്ചരിക്കുന്നതുമാണ്, പക്ഷെ അർക്കിയകളുടെ ഫ്ലജല്ലം മൂലത്തിൽ ഉപശാഖ കൂട്ടിച്ചേരുന്നത്.[68]
ചയാപചയം[തിരുത്തുക]
അർക്കിയ പലതരത്തിലുള്ള ഊർജ്ജഉറവിടങ്ങൾ ഉപയോഗിച്ച് വ്യത്യസ്ത തരത്തിൽ രാസപ്രവർത്തനങ്ങൾ നടത്തി ചയാപചയം നടത്തുന്നു. കാർബൺ അല്ലെങ്കിൽ ഊർജ്ജ ഉറവിടങ്ങളെ അടിസ്ഥാനമാക്കി ഈ രാസപ്രവർത്തനങ്ങളെ ന്യൂട്രീഷൻ ഗ്രൂപ്പുകളായി തരംതിരിച്ചിരിക്കുന്നു. ചില അർക്കിയകൾ അമോണിയ പോലുള്ള (ഇവ ലിഥോട്രോപ്സ് ആണ്) ഇനോർഗാനിക്ക് സംയുക്തങ്ങളിൽ നിന്നും ഊർജ്ജോല്പാദനം നടത്തുന്നു. നൈട്രിഫൈയറുകൾ, മെഥനോജെൻസ്, അനൈറോബിക്ക് മീഥേൻ ഓക്സിഡേർസ് തുടങ്ങിയവ ഇതിലുൾപ്പെടുന്നു.[69] ഈ രാസപ്രവർത്തനങ്ങളിൽ മൂലകങ്ങൾ ഇലക്ട്രോൺ കൈമാറ്റം ചെയ്യുന്നതുവഴി ഊർജ്ജം സ്വതന്ത്രമാവുകയും കോശപ്രവർത്തനങ്ങൽക്ക് ഈ ഊർജ്ജം ഉപയോഗിക്കുകയും ചെയ്യുന്നു. ഒന്ന് ഇലക്ട്രോൺ ദാതാവായും മറ്റേത് സ്വീകർത്താവായും വർത്തിക്കുന്നു. യൂക്കാരിയോട്ടകളുടെ കോശത്തിലെ മൈറ്റോകോണ്ഡ്രിയത്തിലെ അടിസ്ഥാന പ്രവർത്തനം പോലെ ഈ സ്വതന്ത്ര ഊർജ്ജം ക്രോമിയോസ്മോസിസ് വഴി അഡിനോസിൻ ട്രൈഫോസ്ഫേറ്റ് (ATP) ഉണ്ടാക്കുന്നു.[70]
മറ്റുചില അർക്കിയാകൂട്ടങ്ങൾ സൂര്യപ്രകാശം ഊർജ്ജോല്പാദനത്തിനു നേരിട്ടുപയോഗിക്കുന്നു (ഇവ ഫോട്ടോട്രോപ്സ്). എന്നിരുന്നാലും ഓക്സിജൻ നിർമ്മിക്കുന്ന തരത്തിലുള്ള പ്രകാശവിശ്ലേഷണം ഈ ജീവജാലങ്ങളിൽ നടക്കുന്നില്ല.[70] എല്ലാ ജീവജാലങ്ങളിലും പല അടിസ്ഥാന ചയാപചയവ്യവസ്ഥകളും കാണുന്നു, ഉദാഹരണത്തിന് അർക്കിയകൾ ഭേദഗതി വന്ന ഗ്ലൈക്കോസിസ്, മുഴുവനോ ഭാഗികമോ ആയ സിട്രിക്ക് ആസിഡ് സൈക്കിളും ഉപയോഗിക്കുന്നു.[71] മറ്റു ജീവജാലങ്ങലുമായുള്ള ഈ സമാനതകൾ പുരാതന ജീവികളിൽ പ്രതിഫലിപ്പിക്കുന്നത് ഉയർന്ന ക്ഷമതയെ ആണ്.[72]
| പോഷക രീതികൾ | ഊർജ്ജ സ്രോതസ്സുകൾ | കാർബൺ സ്രോതസ്സുകൾ | ഉദാഹരണം |
|---|---|---|---|
| ഫോട്ടോട്രോപ്പുകൾ | സൂര്യപ്രകാശം | ഓർഗാനിക്ക് സംയുക്തങ്ങൾ | ഹാലോബാക്റ്റീരിയ |
| ലിഥോട്രോപ്പുകൾ | ഇനോർഗാനിക്ക് സംയുക്തങ്ങൾ | ഓർഗാനിക്ക് സംയുക്തങ്ങൾ അല്ലെങ്കിൽ കാർബൺ ഫിക്സിങ്ങ് | ഫെറോലോബസ്, മെഥനോബാക്റ്റീരിയ അല്ലെങ്കിൽ പൈറോലോബസ് |
| ഓർഗാനോട്രോപ്പുകൾ | ഓർഗാനിക്ക് സംയുക്തങ്ങൾ | ഓർഗാനിക്ക് സംയുക്തങ്ങൾ അല്ലെങ്കിൽ കാർബൺ ഫിക്സിങ്ങ് | പൈറോകോക്കസ്, സൾഫോലോബസ് അല്ലെങ്കിൽ മെഥനോസാർസിനൽസ് |

ചില യൂക്കാരിയോട്ടകൾ മെഥനോജനുകളാണ് ഇവ ചതുപ്പുനിലം പോലുള്ള വായുവില്ലാത്ത സ്ഥലങ്ങളിൽ കാണപ്പെടുന്നു. ഈ ചയാപചയം വളരെ പുരാതനമാണ്, ഒരുപക്ഷേ ആദ്യ സ്വതന്ത്ര ജീവജാലം മെഥനോജനുകളാകാൻ സാധ്യത ഏറെയാണ്.[74] ഒരു പൊതു രാസപ്രവർത്തനം ഉൾപ്പെട്ടിട്ടുള്ള കാർബൺ ഡൈ ഓക്സൈഡ് ഇലക്ട്രോൺ സ്വീകർത്താവായി വർത്തിച്ച് ഹൈഡ്രജനെ ഓക്സിഡൈസ് ചെയ്യുന്നു. മെഥനോഫ്യൂറാൻ കോഎൻസൈം M തുടങ്ങി, അർക്കിയകളിൽ മാത്രമുള്ള പല സഹരാസാഗ്നികൾ മെഥനോജെനിസിസ് പ്രക്രിയയിൽ ഉൾപ്പെടുന്നു.[75] മറ്റ് ഓർഗാനിക്ക് വസ്തുക്കളായ ആൽക്കഹോൾ, അസറ്റിക് ആസിഡ്, ഫോർമിക്ക് ആസിഡ് തുടങ്ങിയവ മറ്റ് ഇലക്ട്രോൺ സ്വീകർതൃ മാർഗ്ഗമായി മെഥനോജെനുകൾ ഉപയോഗിക്കുന്നു. ഈ പ്രവർത്തനങ്ങൾ ഗട്ട്-ഡ്വള്ളിങ്ങ് അർക്കിയയിൽ സാധാരണമാണ്, ബയോഗ്യാസുണ്ടാക്കുന്ന സൂക്ഷ്മജീവികളിൽ മിക്കവയിലും ഈ പ്രക്രിയയാണ് നടക്കുന്നത്.[76]
മറ്റ് അർക്കിയകൾ അന്തരീക്ഷത്തിലെ CO
2 കാർബൺ സ്രോതസ്സായി ഉപയോഗിക്കുന്നു, ഈ പ്രവർത്തനത്തിനെ കാർബൺ ഫിക്സിങ്ങ് എന്നു പറയുന്നു. ഈ പ്രവർത്തനം ഒന്നുകിൽ ഭേദഗതിവരുത്തിയ കാൽവിൻ സൈക്കിളോ[77] അല്ലെങ്കിൽ അടുത്തിടെ കണ്ടെത്തിയ ചയാപചയ വ്യവസ്ഥയായ 3-ഹെഡ്രോക്സിപ്രൊപ്പിനേറ്റ്/4-ഹൈഡ്രോക്സിബ്യൂറ്റിറേറ്റ് സൈക്കിൾ ഉപയോഗിക്കുന്നു.[78] ക്രെനാർക്കിയോട്ട വിപരീത ക്രെബ്സ് സൈക്കിളും യൂക്കാരിയോട്ടകൾ റിഡക്ടീവ് അസൈറ്റിൽ-CoA പാത്ത്വേ ഉപയോഗിക്കുന്നു.[79] കാർബൺ ഫിക്സിങ്ങിനുള്ള ഊർജ്ജം നൽകുന്നത് ഇനോർഗ്ഗാനിക്ക് ഊർജ്ജസ്രോതസ്സുകളിൽ നിന്നാണ്. അറിയപ്പെടുന്ന ഒരു അർക്കിയകളും പ്രകാശസംശ്ലേഷണം നടത്തുന്നില്ല.[80] അർക്കിയകൾ ഊർജ്ജം സ്വീകരിക്കുന്ന വഴികൾ വ്യത്യസ്തമാണ്, നൈട്രോസോപുമിലാലെസ്[81][82] അമോണിയയുടെ ഓക്സിഡേഷൻ നടത്തുന്നു, ഓക്സിജൻ അല്ലെങ്കിൽ മെറ്റൽ അയോൺ ഇലക്ട്രോൺ സ്വീകർത്താവായി ഉപയോഗിച്ച് സൾഫോലോബസ് ഹൈഡ്രജൻ സൾഫൈഡുകളുടെയോ സൾഫർ മൂലകത്തിന്റെയോ ഓക്സിഡേഷൻ നടത്തി ഇതു സാധ്യമാക്കുന്നു.[70]
ഫോട്ടോട്രോഫിക്ക് അർക്കിയ പ്രകാശം ATP രൂപത്തിലുള്ള രാസോർജ്ജം നിർമ്മിക്കുവാൻ ഉപയോഗിക്കുന്നു. ഹാലൊബാക്റ്റീരിയ, പ്രകാശപ്രേരിതമായ പമ്പുപോലുള്ള ബാക്റ്റീരിയോഹൈഡ്രോഫിൻ, ഹാലൊഡ്രോഫിൻ പ്ലാസ്മാസ്തരത്തിനപ്പുറത്തേക്ക് പായിച്ച് അയോൺ ഗ്രേഡിയന്റുണ്ടാക്കുന്നു. ഈ ഇലക്ട്രോകെമിക്കൽ ഗ്രേഡിയന്റ് പിന്നീട് ATP സിന്തസീസ് വഴി ATP യാക്കിമാറ്റുന്നു.[40] ഈ പ്രക്രിയ ഫോട്ടോഫോസ്ഫോറിലേഷൻ പ്രക്രിയയുടെ വകഭേദമാണ്. പ്രോട്ടീന്റെ ഉള്ളിൽ മധ്യഭാഗത്തുള്ള റെറ്റിനോൾ കോഫാക്ടറിലുണ്ടാകുന്ന മാറ്റമാണ് പ്ലാസ്മാസ്തരത്തിനപ്പുറത്തേക്ക് പായിക്കനുള്ള കഴിവ് ആശ്രയിച്ചിരിക്കുന്നത്.[83]
ജനിതകം[തിരുത്തുക]
അർക്കിയകൾക്ക് വൃത്താകൃതിയിലുള്ള ഒരു ക്രോമോസോമാണുള്ളത്,[84] വലിപ്പം 5,751,492 അടിസ്ഥാന ജോഡികൾ വരെ ആകാം മീഥനോസക്രിന അസെറ്റിവൊറൻസിൽ,[85] ഇതാണ് അറിയപ്പെടുന്ന അർക്കിയാ ജീനോം. നാനോഅർക്കിയം ഇക്യുറ്റിയൻസിന് ഇതിന്റെ പത്തിലൊന്ന് വലിപ്പമുള്ള ജീനോമാണുള്ളത്, ഇതാണ് അറിയപ്പെടുന്നതിലെ ഏറ്റവും ചെറിയ ജീനോം. ഇവ 573 പ്രോട്ടീൻ-അലേഖനംചെയ്ത ജീനുകൾ മാത്രമേ ഉള്ളൂ.[86] പ്ലാസ്മിഡുകൾ എന്നു പറയുന്ന ഡി.എൻ.എ യുടെ സ്വതന്ത്ര ശകലങ്ങളും അർക്കിയകളിൽ കാണപ്പെടുന്നു. ഭൗതികസ്പർശം മൂലം പ്ലാസ്മിഡുകളുടെ കൈമാറ്റം സാധ്യമാകാം, ഇവ ബാക്റ്റീരികളുടെ കോഞ്ചുഗേഷൻ പ്രവർത്തനത്തോട് സമാനത പുലർത്തുന്നു.[87][88]

അർക്കിയ മറ്റു വൈറസ്സുകളിൽ നിന്നും ബന്ധമില്ലാത്തതും അസാധാരണ ആകൃതിയിലുള്ള (ബോട്ടിൽ, ഹുക്ക്ഡ് റോഡ്, ടെറാഡ്രോപ്സ്) ഇരട്ട ഇഴകളുള്ള ഡി.എൻ.എ വൈറസ്കളുടെ സംക്രമണം ഉണ്ടാകാം.[90] തെർമോഫിലിക്സിൽ ഈ വൈറസുകളെക്കുറിച്ച് വിശദമായി പഠിച്ചിട്ടുണ്ട്, പ്രത്യേകിച്ച് സൾഫോലൊബലസ്, തെർമമോപ്രൊറ്റെലെസ് ക്രമങ്ങളിൽ.[91] ഹാലോഫിലിക്ക് അർക്കിയയിൽ സംക്രമിക്കുന്ന ഒരിഴ മാത്രമുള്ള ഡി.എൻ.എ വൈറസുകളെ 2009-ൽ കണ്ടെത്തി.[92] വൈറസ്സുകളുടെ ജീനുകളുമായി ബന്ധമുള്ള റിപീറ്റഡ് ഡി.എൻ.എ അനുക്രമത്തിലെ ആർ.എൻ.എ കൈകടത്തൽ ഈ വൈറസുകൾക്കെതിരെയുള്ള പ്രതിരോധം സാധ്യമാക്കിയേക്കാം.[93][94]
അർക്കിയ സാധാരണയായി ബാക്റ്റീരിയയിൽ നിന്നും യൂക്കാരിയോട്ടകളിൽ നിന്നും വ്യത്യസ്തമാണ്, ഏതെങ്കിലും ഒരു അർക്കിയയുടെ ജീനോം കൊണ്ട് 15% പ്രോട്ടീൻ എങ്കോഡിങ്ങ് നടന്നാൽ സാമ്രാജ്യം തികച്ചും വ്യത്യസ്തമായിരിക്കും, മിക്ക നിരുപമ ജീനുകൾക്കും അറിയപ്പെടുന്ന പ്രവർത്തനങ്ങളുമില്ല.[95] ചില നിരുപമ പ്രോട്ടീനുകളുടെ പ്രവർത്തനം മനസ്സിലാക്കിയിട്ടുണ്ട്, ഇവയിൽ മിക്കവയും യൂറിയാർക്കിയയും ഇവ മെഥനോജനിസിസിൽ പങ്കുള്ളവയുമാണ്. അർക്കിയ, ബാക്ടീരിയ, യൂക്കാരിയോട്ടകൾ എന്നിവ പങ്കിടുന്ന പ്രോട്ടീനുകൾ ഒരു പൊതു കേന്ദ്ര കോശപ്രവർത്തനം ബന്ധപ്പെടുന്നത് ട്രാൻസ്ക്രിപ്ഷൻ, ട്രാൻസിലേഷൻ, ന്യൂക്ലിയോറ്റൈഡ് മെറ്റബോളിസം എന്നിവയോടാണ്.[96] ജീനുകളുടെ പ്രവർത്തനങ്ങളാണ് മറ്റു അർക്കിയകളെ പ്രത്യേകതയുള്ളതാക്കുന്നത്, ചയാപചയത്തിൽ സഹായിക്കുന്ന രാസാഗ്നികളിൽ ഉൽപ്രേരകങ്ങൾ മെറ്റബോളിക് ഓപറോണുകൾ ആഖ്യായിക മാറ്റങ്ങൾ വരുത്തുന്നു, മാത്രമല്ല tRNA ജീനുകളിലും അമിനോസിൽ tRNA സിന്തസീസിലും.[96]
അർക്കിയയിലെ ട്രാൻസ്ക്രിപ്ഷൻ ട്രാർസലേഷൻ ബാക്ടീരിയയേക്കാൾ യൂക്കാരിയോട്ടകളുമായി സാമ്യപ്പെടുന്നു, അർക്കിയയിലെ RNA പോളിമറുകളും റൈബോസോമുകളും യൂക്കാരിയോട്ടക്ലിലേതുപോലെ കാണുന്നു.[84] അർക്കിയകൾക്ക് ഒരേ രീതിയിലുള്ള RNA പോളിമർ മാത്രമേ ഉള്ളൂ, ഇവയുടെ ആകൃതിയും ട്രാൻസ്ക്രിപ്ഷൻ പ്രവർത്തനവും യൂക്കാരിയോട്ടകളുടെ ആർ.എൻ.എ പോളിമർ II വളരെ സാമ്യമുള്ളതാണ്, സമാനമായ പ്രോട്ടീൻ അസബ്ലികൾ ആർ.എൻ.എ പോളിമറുകളെ ജീൻ പരിപോഷികളാക്കുന്നു.[97] എന്നിരുന്നാലും മറ്റു അർക്കിയൻ ട്രാൻസ്ക്രിപ്ഷൻ ബാക്ടീരിയകളോട് സാമ്യമുള്ളവയാണ്.[98] ട്രാൻസ്ക്രിപ്ഷനുശേഷമുള്ള പരിഷ്ക്കരിക്കൽ യൂക്കാരിയോട്ടകളേപ്പോലെയാണ്, മിക്ക അർക്കിയൻ ജീനുകളിൽ ഇൻട്രോൺ കാണുന്നില്ല, എന്നാൽ കൈമാറ്റം ചെയ്യുന്ന ആർ.എൻ.എ കളിലും, റൈബോസോമൽ ആർ.എൻ.എ ജീനുകളിലും ഇൻട്രോണുകൾ കാണപ്പെടുന്നു,[99] ചില പ്രോട്ടീൻ എൻകോഡിങ്ങ് ജീനുകളിൽ ഇൻട്രോണുകൾ ഉണ്ടാകാം.[100][101]
പ്രത്യുൽപ്പാദനം[തിരുത്തുക]
ഇവ അലൈംഗിക പ്രത്യുൽപ്പാദനം വഴി വംശവർദ്ധന നടത്തുന്നു, ദ്വിഖണ്ഡനം, ബഡ്ഡിംഗ്, ഫ്രാഗ്മെന്റേഷൻ, തുടങ്ങിയരീതികൾ അവലംബിക്കുന്നു, അതുകൊണ്ട് തന്നെ അർക്കിയകൾ ഒന്നിലധികം സ്പീഷീസിൽ ഇവ ഉണ്ടെങ്കിലും, അവയ്ക്കെല്ലാം ഒരേ ജനിതകഊറ ഉണ്ടായിരിക്കും.[40] കോശ സൈക്കിളിൽ കോശവർദ്ധന നിയന്ത്രിതമാണ്, കോശത്തിലെ ക്രോമോസോം രണ്ട് ശിശു ക്രോമോസോമായി വേർപെട്ടശേഷം കോശം രണ്ടായിത്തീരുന്നു.[102] വിശദവിവരങ്ങൾ സൾഫോലോബസ് ജനുസ്സുകൾക്ക് മാത്രമേ ലഭിച്ചിട്ടുള്ളൂ, എന്നാൽ ഈ കോശ സൈക്കിൾ ബാക്റ്റീരിയ യൂക്കാരിയോട്ടകളോടും സാമ്യമുള്ളതാണ്. DNA പോളിമറേസ് ഉപയോഗിച്ച് പല ഭാഗങ്ങളിൽ നിന്ന് ക്രോമോസോമുകൾ വിഭജിച്ചുതുടങ്ങുന്നു, ഇവ യൂക്കാരിയോട്ട രാസാഗ്നിയോട് വളരെ സാമ്യമുണ്ട്.[103] എന്നിരുന്നാലും കോശവിഭജനത്തിനെ നിയന്ത്രിക്കുന്ന FtsZ പോലുള്ള പ്രോട്ടീനുകൾ ഉണ്ടാക്കുന്ന കോശത്തിനു ചുറ്റുമുള്ള ചുരുങ്ങുന്ന വളയം, കോശമധ്യത്തിലുള്ള നിർമ്മാണഘടനയിലെ കള്ളികളും ബാക്ടീരിയകളുടേതുപോലെയാണ്.[102]
ബാക്റ്റീരിയയും യൂക്കാരിയോട്ടകളും ഉണ്ടാക്കുന്നെങ്കിലും അർക്കിയ ബീജാണു ഉണ്ടാക്കുന്നില്ല.[104] ചില സ്പീഷിസിലുള്ള ഹാലോഅർക്കിയ ഫീനോടിപ്പിക് സ്വിച്ചിങ്ങ് വഴി പല തരം കോശങ്ങൾ വളർത്തുന്നു, കട്ടികൂടിയ കോശസ്തരത്തോടുകൂടിയവയും ഇതിൽപെടുന്നു. ഇവ ഓസ്മോട്ടിക് ആഘാതത്തിൽ നിന്നും സംരക്ഷിക്കുന്നു. കുറഞ്ഞ ഉപ്പ് നിലനിർത്തി ഇവയ്ക്ക് ജലത്തിൽ നിലനിൽക്കാൻ സാധിക്കുന്നതു ഇതുമൂലമാകാം, എന്നിരുന്നാലും ഇതൊന്നും പ്രത്യുല്പാദനവുമായി ബന്ധമില്ലാത്തതാണ്, എന്നാൽ പുതിയ ആവാസവ്യവസ്ഥക്ക് അനുയോജ്യമാക്കുന്നു.[105]
പരിതഃസ്ഥിതി[തിരുത്തുക]
ജീവസാഹചര്യം[തിരുത്തുക]
പലതരത്തിലുള്ള ജീവസാഹചര്യങ്ങളിലും അർക്കിയകൾ കണ്ടുവരുന്നു, ഭൂമിയിലെ ആവാസവ്യവസ്ഥയിലെ വലിയ ഒരു ഭാഗംതന്നെ അർക്കിയകളാണ്.[7] ഭൂമിയുടെ ജൈവപിണ്ഡത്തിന്റെ 20% ത്തോളം വരും ഇവ.[106] ഘോര ജീവസാഹചര്യങ്ങളിൽ നിന്നാണ് ആദ്യം അർക്കിയകളെ കണ്ടെത്തിയത്.[69] ചില അർക്കിയകൾ ഉയർന്ന താപനിലകളിൽ നിലനിൽക്കുന്നു, എണ്ണക്കിണറുകൾ, ഗൈസറുകൾ പോലുള്ള 100 °C (212 °F) നു മുകളിൽ താപനിലയുള്ള സ്ഥലങ്ങളിൽ ഇവയെ കണ്ടെത്തിയിരിക്കുന്നു. മറ്റു പൊതു ജീവസാഹചര്യങ്ങൾ തണുപ്പു കൂടിയതും, ഉപ്പുകൂടിയതോ, അമ്ലാവസ്ഥയുള്ളതോ, ക്ഷാരാവസ്ഥയുള്ളതോ ആയ ജലത്തിൽ കാണപ്പെടുന്നു. എന്നിരുന്നാലും മീസോഫീലുകളായ അർക്കിയകൾ അഘോരസാഹചര്യങ്ങളായ ചതുപ്പുനിലം, ഓടകൾ, സമുദ്രങ്ങൾ, മണ്ണ് എന്നിടങ്ങളിലും കാണപ്പെടുന്നു.[7]

എക്സ്ട്രീമോഫൈൽ (ഘോര ജീവസാഹചര്യങ്ങളിൽ വളരുന്നവ) അർക്കിയകൾ നാലു പ്രധാന ശരീരശാസ്ത്രകൂട്ടങ്ങളായി കാണുന്നു, ഇവ ഹാലോഫൈൽ, തെർമോഫൈൽ, ആൽക്കലിഫൈൽ, അസിഡോഫൈൽ എന്നിവയാണ്.[107] ഇവ വർഗ്ഗീകരിച്ചിരിക്കുന്നത്, ഫൈലങ്ങളായല്ല, ചില അർക്കിയകൾ രണ്ടിലധികം കൂട്ടങ്ങളിലും കാണപ്പെടാം. എന്നിരുന്നാൽ പ്രയോജനമേറിയ വർഗ്ഗീകരണത്തിന്റെ തുടക്കമാണ് ഇത്. ഹാലോബാക്റ്റീരിയം ഉൾപ്പെടെ ഹാലോഫൈലുകൾ വളരെ ഘോരമായ ലവണ പരിതഃസ്ഥിതി ഉള്ള സ്ഥലങ്ങളിലാണ് വളരുന്നത്, ഉപ്പ്വെള്ളമുള്ള കായലുകളിൽ വളരുന്ന ഇവ മറ്റ് സമാന ജീവികളായ ബാക്ടീരിയെക്കാൾ 20–25% കൂടുതലാണ്.[69] തെർമോഫൈൽ 45 °C (113 °F) ൽ കൂടുതൽ ഊഷ്മാവുള്ള സ്ഥലങ്ങളിലാണ് ഉത്തമമായി വളരുന്നത്, എന്നാൽ ഹൈപ്പർതെർമോഫൈലുകൾ താപനീരുറവകളിലെ 80 °C (176 °F) ൽ അധികമായ ഊഷ്മാവിൽ സുഖമായി വളരുന്നു.[108] അർക്കിയയായ മെഥനോഫിറസ് ക്ൻഡ്ലേറി സ്റ്റ്രെയിൻ 116 വളരുന്നത് 122 °C (252 °F) ഊഷ്മാവിലാണ്, കണ്ടെത്തിയതിൽ ഇതാണ് ഏതെങ്കിലും ജീവജാലം വളരുന്ന ഏറ്റവും കൂടിയ താപനില.[109] മറ്റ് അർക്കിയകൾ വളരെ അമ്ലാവസ്ഥയിലോ ആൽക്കലൈൻ സാഹചര്യങ്ങളിലോ കാണുന്നു.[107] ഉദാഹരണത്തിനായി അസിഡോഫൈൽ അർക്കിയകളിൽ ഒന്നായ പൈക്രോഫിലസ് ടൊറിഡസ് വളരുന്നത് പി.എച്ച് 0 യിലാണ്, ഇത് 1.2 മോളാർ സൾഫ്യൂരിക് അമ്ലത്തിനു തുല്യമാണ്.[110]
ഈ കടുത്ത ജീവസാഹചര്യങ്ങളിൽ വർത്തിക്കുന്നതിനാൽ ഇവ ഭൂമിക്കു പുറത്തുനിന്നുവന്ന ജീവികൾ ആണെന്നു ചിന്തിപ്പിക്കുന്നു.[111] ചില എക്സ്ട്രീമോഫൈൽ ചൊവ്വയിലുള്ളതിൽ നിന്നു വ്യത്യസ്തമല്ല,[112] അതിനാൽ സൂക്ഷ്മജീവികൾ ഉൽക്കാപതനത്താൽ ഗ്രഹങ്ങളിൽ കൈമാറ്റം ചെയ്യപ്പെട്ടതാകാം.[113]
അടുത്തിടെ നടന്ന പഠനങ്ങൾ പറയുന്നത്, പരിമിത, അതി താപനിലകളിൽ വളരുന്നവ മാത്രമല്ല വളരെ താഴ്ന്ന താപനിലയിലും ഇവയെ അധികമായി കാണുന്നു. ഉദാഹരണത്തിന്, അർക്കിയകൾ ധ്രുവപ്രദേശ സമുദ്രങ്ങളിലെ വളരെ കുറഞ്ഞ താപനിലയിലും കാണുന്നു.[114] വളരെ അധികം അർക്കിയകളെ പ്ലാങ്ടൺ സമൂഹങ്ങളുടെ കൂടെ (പൈക്കോ പ്ലാങ്ടണിന്റെ കൂടെ) പരിമിത സാഹചര്യങ്ങളുള്ള ലോക സമുദ്രങ്ങളിൽ ദൃശ്യമാണ്.[115] ഈ അർക്കിയകൾ എണ്ണത്തിൽ വളരെ കൂടുതലായി (സൂക്ഷ്മ ജൈവപിണ്ഡത്തിന്റെ 40% ത്തോളം) കാണുന്നു, എന്നിരുന്നാലും മിക്ക സ്പീഷീസും വേർതിരിച്ചെടുത്ത് കൾച്ചർ ചെയ്ത് പഠനവിധേയമാക്കിയിട്ടില്ല.[116] അർക്കിയകൾ സമുദ്ര പരിതഃസ്ഥിതികളെ എത്രത്തോളം സ്വാധീനിക്കുന്നു എന്നുള്ളത് വികസിതമല്ലാത്ത അറിവുകളാണ്, അതിനാൽ ജൈവരാസ ചക്രങ്ങളിൽ ഇവയ്ക്കുള്ള പങ്ക് കണ്ടുപിടിച്ചിട്ടില്ല.[117] ചില സമുദ്ര റീനാർക്കിയകൾ നെട്രജൻ സ്ഥിരീകരണം നടത്തുവാൻ കഴിവുള്ളവയാണ്, സമുദ്രതട നൈട്രജൻ ചക്രത്തിൽ സ്വാധീനം ചെലുത്തുവാൻ ഇവയ്ക്കു കഴിയുമായിരിക്കാം,[118] മാത്രമല്ല സമുദ്ര റീനാർക്കിയകൾ മറ്റു ഊർജ്ജരൂപങ്ങൾ ഉപയോഗപ്പെടുത്തുന്നതിനും സാധ്യതയുണ്ട്.[119] വലിയ ഒരു കൂട്ടം അർക്കിയകൾ സമുദ്ര തട അടിച്ചിലിൽ കാണുന്നു, സമുദ്ര തടങ്ങൾക്ക് ഒരു മീറ്റർ താഴെയായി ഈ ജീവജാലങ്ങൾ കഴിഞ്ഞുകൂടുന്നു.[120][121]
ജൈവരാസ ചംക്രമണത്തിലെ പങ്ക്[തിരുത്തുക]
അർക്കിയ കാർബൺ, നൈട്രജൻ, സൾഫർ തുടങ്ങിയ മൂലകങ്ങളുടെ പുനർചംക്രമണം പല ആവാസവ്യവസ്ഥയിൽ നടത്തുന്നു. ഈ പ്രവൃത്തി ആവാസവ്യവസ്ഥയുടെ കൃത്യനിർവ്വഹണത്തിൽ മർമ്മപ്രധാനമായ പങ്ക് വഹിക്കുന്നു, മനുഷ്യനിർമ്മിതമായ മാറ്റങ്ങളും മലിനീകരണത്തിലും ഇവയ്ക്ക് സ്വാധീനം ചെലുത്താം.
നെട്രജൻ ചക്രത്തിലെ പല പടികളും അർക്കിയ്കൾ നടത്തുന്നു. ഈ പ്രവൃത്തി നെട്രേറ്റിനെ അടിസ്ഥാനമാക്കിയ ശ്വസനം, ഡീനൈട്രിഫിക്കേഷൻ തുടങ്ങി നെട്രജനെ ആവാസവ്യവസ്ഥയിൽനിന്നും മാറ്റുന്നതും, നെട്രേറ്റ് ലയനം നെട്രജൻ സ്ഥിരീകരണം തുടങ്ങി നെട്രജനെ ആവാസവ്യവസ്ഥയിലേക്കെത്തിക്കുന്നതും ഉൾപ്പെടും.[122][123] അമോണിയയുടെ ഓക്സിഡേഷനിലുള്ള അർക്കിയകളുടെ പങ്ക് അടുത്തിടെ കണ്ടെത്തി. ഈ പ്രതിപ്രവർത്തനം സമുദ്രങ്ങളിൽ പ്രധാനപ്പെട്ടവയാണ്.[124][125] മണ്ണിലെ അമോണിയ ഓക്സിഡേഷനിലും അർക്കിയകൾ നിർണ്ണായകമാകുന്നു. ഇവ നൈട്രൈറ്റ് ഉണ്ടാക്കുന്നു, പിന്നീട് മറ്റ് സൂക്ഷ്മജീവികൾ ഓക്സിഡൈസ് ചെയ്ത് നൈട്രേറ്റ് ആക്കുന്നു. സസ്യങ്ങളും മറ്റു ജീവികളും ഇത് ഉപഭോഗിക്കുന്നു..[126]
സൾഫർ ചക്രത്തിൽ അർക്കിയകൾ വളരുന്നത് പാറകളിലെ സൾഫർ സംയുക്തങ്ങളെ ഓക്സിഡൈസ് ചെയ്താണ്, ഇവ മറ്റ് ജീവജാലങ്ങൾ ഉപയോഗപ്പെടുത്തുന്നു. എന്നിരുന്നാലും, സൾഫോലോബസ് പോലുള്ള ഇത്തരം അർക്കിയകൾ രാസപ്രവർത്തനത്തിന്റെ ഫലമായി സൾഫ്യൂരിക്ക് അമ്ലം അവശേഷിപ്പിക്കുന്നു, ഇങ്ങനെയുള്ളവ ഉപയോഗശൂന്യമായ ഖനികളിൽ അമ്ലശോഷനത്തിനിടയാക്കുകയോ പരിസ്ഥിതി നാശങ്ങളോ വരുത്തിവെയ്ക്കും.[127]
കാർബൺ ചക്രത്തിൽ അടിച്ചിലുകൾ, ചതുപ്പുകൾ, ഓടകൾ തുടങ്ങിയ വായുവില്ലാത്ത സാഹചര്യങ്ങളിൽ സൂക്ഷ്മ ജീവികൾ ഓർഗാനിക് അവശിഷ്ടങ്ങളുടെ വിഘടനം നടത്തുമ്പോൾ മെഥനോജെനുകൾ ഹൈഡ്രജൻ നീക്കം ചെയ്യുന്നു.[128] എന്നിരുന്നാലും ഭൗമാന്തരീക്ഷത്തിൽ മീഥൈൻ കൂടുതലായുള്ള ഹരിതഗൃഹ വാതകമാണ്, ലോകത്തിന്റെ 18% ഇതാണ്.[129] കാർബൺ ഡൈ ഓക്സൈഡിനേക്കാൾ 25 മടങ്ങ് ഹരിതഗൃഹപ്രഭാവശേഷി കൂടുതലുള്ളവയാണിത്.[130] മെഥനോജെനുകളാണ് അന്തരീക്ഷത്തിലെ മീഥൈന്റെ പ്രധാന ഉറവിടം, ഇവയാണ് ലോകത്തിലെ വാർഷിക മീഥൈൻ പുറംതള്ളലിന്റെ കാരണക്കാർ.[131] ഇതിന്റെ പരിണതഫലമായി, അർക്കിയകളാണ് ഹരിതഗൃഹ വാതകങ്ങൾക്കും ആഗോളതാപനത്തിനും കാരണമാകുന്നത്.
മറ്റ് ജീവജാലങ്ങളുമായുള്ള സഹവർത്തിത്വം[തിരുത്തുക]

അർക്കിയകളും മറ്റു ജീവികളും പരസ്പരപ്രവർത്തനം അന്യോന്യമായതോ പരപോഷണത്തിലൂടെയോ സാധ്യമാക്കുന്നു. എന്നാൽ അറിയപ്പെടുന്ന അർക്കിയകൾ രോഗാണുക്കളോ പരാദങ്ങളോ ഇല്ല.[132][133] എന്നാൽ, ചില മെഥനോജെൻ സ്പീഷീസുകൾ വായിലുള്ള ബാധയായി ഇവ കാണുന്നു,[134][135] പക്ഷെ നാനോഅർക്കിയം ഇക്വിറ്റൻസ് മറ്റ് അർക്കിയൻ സ്പീഷികളിൽ പരാദമായി കാണപ്പെടുന്നു, അതുകൊണ്ട് തന്നെ ഇവ ജീവിക്കുന്നതും വംശവർദ്ധന നടത്തുന്നതും ഗിനികോക്കസ് ഹോസ്പിറ്റലിസിന്റെ കോശത്തിനുള്ളിൽ തന്നെയാണ്[136], എന്നാൽ ആതിഥേയനു യാതൊരു ഗുണവും ചെയ്യുന്നില്ല.[137]
ചുരുക്കിപ്പറഞ്ഞാൽ അർക്കിയൽ റിച്ച്മണ്ട് മൈൻ അസിഡോഫിലിക്ക് നനോർഗാനിസം അമ്ല ഖനികളിലെ ചാലുകളിലെ ബയോഫിലിമുകളിൽ അപ്പോഴപ്പോൾ മറ്റ് അർക്കിയൻ കോശങ്ങളിൽ ചേരുന്നു.[138] എന്നിരുന്നാലും ഇവയുടെ സ്വഭാവം ഇപ്പോഴും വ്യക്തമല്ല.
സാങ്കേതിക വ്യവസായിക പ്രാമുഖ്യം[തിരുത്തുക]
എക്സ്ട്രിമോഫൈൽ അർക്കിയകൾ കൂടിയ ഊഷ്മാവിനോടോ, അമ്ല ക്ഷാര അവസ്ഥകളോടോ പ്രതിരോധമുള്ളവയാണ്, ഈ പരിതഃസ്ഥിതികളിൽ നിലനിൽക്കുവാൻ ഉതകുന്ന രാസാഗ്നികളുടെ ഉറവിടമാണിവ.[139][140] ഈ രാസാഗ്നികൾക്ക് ഉപയോഗങ്ങളേറെയാണ്. ഉദാഹരണത്തിന് തന്മാത്രാ ജീവശാസ്ത്രത്തിൽ ഗവേഷണത്തിനായി ഡി.ൻ.എ ക്ലോണിംഗ് നടത്തുവാൻ ലളിതവും വേഗത്തിലുമുള്ള പളിമറേസ് ചെയിൻ പ്രതിപ്രവർത്തനം നടത്തുവാൻ താപമാറ്റങ്ങൾക്ക് വിധേയമാകാത്ത Pfu ഡി.എൻ.എ പോളിമറേസ് പോലുള്ള ഡി.എൻ.എ പോളിമറേസ് ഉപയോഗിക്കുന്നു. വ്യാവസായത്തിൽ, അമിലേസ്, ഗാലക്ടോസിഡേസ്, പുല്ലുലാനസെസ് മറ്റു ചില പൈറൊകോക്കസ് ഉപവർഗ്ഗം എന്നിവ 100 °C (212 °F) മുകളിൽ ഭക്ഷണ സംസ്കരണം നടത്തുവാൻ സഹായിക്കുന്നു, കുറഞ്ഞ ലാക്റ്റോസ് പാല്, പാൽ രസം പോലുള്ള ഭക്ഷണങ്ങൾ ഇതിനുദാഹരണമാണ്.[141] ഓർഗാനിക്ക് ലായനികളിലും, തെർമോഫിലിക് അർക്കിയകളിൽ നിന്നുള്ള രാസാഗ്നികൾ മാറ്റമില്ലാത്തതാണ്, ഇതിനാൽ പ്രകൃതിയോടിണങ്ങിയ ഹരിത രസതന്ത്ര പ്രക്രിയയാൽ ഓർഗാനിക്ക് സംയുക്തം സിന്തസീസ് ചെയ്യാൻ ഉതകുന്നു.[140] ഈ സ്വഭാവം ഘടനാ ജീവശാസ്ത്രത്തിൽ ഉപയോഗിക്കാൻ ഉപകരിക്കുന്നു. പരിണതഫലമായി ഇതേ സ്വഭാവ സവിശേഷത ഉള്ള എക്സ്ട്രിമോഫൈൽ അർക്കിയകളിലെ ബാക്റ്റീരിയ, യൂക്കാരിയോട്ട രാസാഗ്നികൾ ഘടനാ ജീവശാസ്ത്ര പഠനത്തിനുപയോഗിക്കുന്നു.[142]
രാസാഗ്നികളുടെ വിവിധ ഉപയോഗങ്ങളല്ലാതെ, നേരിട്ട് ഈ ജീവജാലത്തിനെ തന്നെ ജൈവസാങ്കേതികവിദ്യയിൽ ഉപയോഗിക്കുന്നത് വികസിച്ചിട്ടില്ല. ഓടകളുടെ സംസ്കരണത്തിനു മെഥനോജെനിക്ക് അർക്കിയകൾ പ്രധാനപങ്കു വഹിക്കുന്നു, ഇവ മറ്റ് ചില സൂക്ഷ്മ ജീവികൾ പോലെ അവായവദഹനം വഴി ജൈവ വാതകം നിർമ്മിക്കുന്നു.[143] സ്വർണ്ണം, കൊബാൾട്ട്, ചെമ്പ് എന്നിവ ഉൾപ്പെടെ അയിരുസംസ്കരണത്തിൽ അയിരിൽ നിന്ന് ലോഹത്തിനെ വേർതിരിക്കുന്നതിന് അസിഡോഫിലിക്ക് അർക്കിയകൾ ഉപകരിക്കും.[144]
ഇവ അയേക്കാവുന്ന പുതിയ ആന്റിബയോട്ടിക്ക് ഗണമാണ്. ഇതിൽ കുറച്ചെണ്ണം ഈ വിശേഷമുള്ളതായി സ്ഥിരീകരിച്ചിട്ടുണ്ടെങ്കിലും നൂറില്പരം ഉണ്ടെന്നു വിശ്വസിക്കുന്നു, പ്രത്യേകിച്ച് ഹാലോഅർക്കിയത്തിലും സൾഫോലോബസിലും.[145] ബാക്റ്റീരിയത്തിൽ നിന്നുള്ള ആന്റിബയോട്ടിക്കുകൾ ഘടനയിൽ വ്യത്യസ്തമാണ്, അതുകൊണ്ട്തന്നെ ആഖ്യായികമായ പ്രവർത്തനങ്ങൾ കാണാം. മാത്രമല്ല, ചിലപ്പോൾ പുതിയ കോശാന്തര ജീൻ പരിവർത്തനം നടത്തുവാൻ അർക്കിയൻ തന്മാത്രാ ജീവശാസ്ത്രത്തിന് കഴിഞ്ഞേക്കാം.[146]
അവലംബം[തിരുത്തുക]
- ↑ Pace NR (2006). "Time for a change". Nature. 441 (7091): 289. Bibcode:2006Natur.441..289P. doi:10.1038/441289a. PMID 16710401.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Staley JT (2006). "The bacterial species dilemma and the genomic-phylogenetic species concept". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1475): 1899–909. doi:10.1098/rstb.2006.1914. PMC 1857736. PMID 17062409.
- ↑ Zuckerkandl E, Pauling L (1965). "Molecules as documents of evolutionary history". J. Theor. Biol. 8 (2): 357–66. doi:10.1016/0022-5193(65)90083-4. PMID 5876245.
- ↑ Woese C, Fox G (1977). "Phylogenetic structure of the prokaryotic domain: the primary kingdoms". Proc Natl Acad Sci USA. 74 (11): 5088–90. Bibcode:1977PNAS...74.5088W. doi:10.1073/pnas.74.11.5088. PMC 432104. PMID 270744.
- ↑ Woese CR, Kandler O, Wheelis ML (1990). "Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya". Proc. Natl. Acad. Sci. U.S.A. 87 (12): 4576–9. Bibcode:1990PNAS...87.4576W. doi:10.1073/pnas.87.12.4576. PMC 54159. PMID 2112744. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ archaea. (2008). In Merriam-Webster Online Dictionary. Retrieved July 1, 2008, from http://www.merriam-webster.com/dictionary/archaea Archived 2015-09-12 at the Wayback Machine.
- ↑ 7.0 7.1 7.2 DeLong EF (1998). "Everything in moderation: archaea as 'non-extremophiles'". Curr. Opin. Genet. Dev. 8 (6): 649–54. doi:10.1016/S0959-437X(98)80032-4. PMID 9914204.
- ↑ Theron J, Cloete TE (2000). "Molecular techniques for determining microbial diversity and community structure in natural environments". Crit. Rev. Microbiol. 26 (1): 37–57. doi:10.1080/10408410091154174. PMID 10782339.
- ↑ Schmidt TM (2006). "The maturing of microbial ecology" (PDF). Int. Microbiol. 9 (3): 217–23. PMID 17061212. Archived from the original (PDF) on 2008-09-11. Retrieved 2012-03-13.
- ↑ Gevers D, Dawyndt P, Vandamme P; et al. (2006). "Stepping stones towards a new prokaryotic taxonomy". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1475): 1911–6. doi:10.1098/rstb.2006.1915. PMC 1764938. PMID 17062410.
{{cite journal}}: Explicit use of et al. in:|author=(help)CS1 maint: multiple names: authors list (link) - ↑ 11.0 11.1 Robertson CE, Harris JK, Spear JR, Pace NR (2005). "Phylogenetic diversity and ecology of environmental Archaea". Curr. Opin. Microbiol. 8 (6): 638–42. doi:10.1016/j.mib.2005.10.003. PMID 16236543.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO. (2002). "A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont". Nature. 417 (6884): 27–8. doi:10.1038/417063a. PMID 11986665.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Barns SM, Delwiche CF, Palmer JD, Pace NR (1996). "Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences". Proc. Natl. Acad. Sci. U.S.A. 93 (17): 9188–93. Bibcode:1996PNAS...93.9188B. doi:10.1073/pnas.93.17.9188. PMC 38617. PMID 8799176. Archived from the original on 2020-05-08. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Elkins JG, Podar M, Graham DE; et al. (2008). "A korarchaeal genome reveals insights into the evolution of the Archaea". Proc. Natl. Acad. Sci. U.S.A. 105 (23): 8102–7. Bibcode:2008PNAS..105.8102E. doi:10.1073/pnas.0801980105. PMC 2430366. PMID 18535141. Archived from the original on 2020-05-27. Retrieved 2012-03-13.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Baker, B.J., Tyson, G.W., Webb, R.I., Flanagan, J., Hugenholtz, P. and Banfield, J.F. (2006). "Lineages of acidophilic Archaea revealed by community genomic analysis. Science". Science. 314 (6884): 1933–1935. Bibcode:2006Sci...314.1933B. doi:10.1126/science.1132690. PMID 17185602.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Baker BJ, Comolli LR, Dick GJ; et al. (2010). "Enigmatic, ultrasmall, uncultivated Archaea". Proc. Natl. Acad. Sci. U.S.A. 107 (19): 8806–11. Bibcode:2010PNAS..107.8806B. doi:10.1073/pnas.0914470107. PMC 2889320. PMID 20421484.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ de Queiroz K (2005). "Ernst Mayr and the modern concept of species". Proc. Natl. Acad. Sci. U.S.A. 102 (Suppl 1): 6600–7. Bibcode:2005PNAS..102.6600D. doi:10.1073/pnas.0502030102. PMC 1131873. PMID 15851674. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
- ↑ Eppley JM, Tyson GW, Getz WM, Banfield JF (2007). "Genetic exchange across a species boundary in the archaeal genus ferroplasma". Genetics. 177 (1): 407–16. doi:10.1534/genetics.107.072892. PMC 2013692. PMID 17603112. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Papke RT, Zhaxybayeva O, Feil EJ, Sommerfeld K, Muise D, Doolittle WF (2007). "Searching for species in haloarchaea". Proc. Natl. Acad. Sci. U.S.A. 104 (35): 14092–7. Bibcode:2007PNAS..10414092P. doi:10.1073/pnas.0706358104. PMC 1955782. PMID 17715057. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Kunin V, Goldovsky L, Darzentas N, Ouzounis CA (2005). "The net of life: reconstructing the microbial phylogenetic network". Genome Res. 15 (7): 954–9. doi:10.1101/gr.3666505. PMC 1172039. PMID 15965028. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Hugenholtz P (2002). "Exploring prokaryotic diversity in the genomic era". Genome Biol. 3 (2): REVIEWS0003. doi:10.1186/gb-2002-3-2-reviews0003. PMC 139013. PMID 11864374. Archived from the original on 2020-03-15. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ↑ Rappé MS, Giovannoni SJ (2003). "The uncultured microbial majority". Annu. Rev. Microbiol. 57: 369–94. doi:10.1146/annurev.micro.57.030502.090759. PMID 14527284.
- ↑ Schopf J (2006). "Fossil evidence of Archaean life" (PDF). Philos Trans R Soc Lond B Biol Sci. 361 (1470): 869–85. doi:10.1098/rstb.2006.1834. PMC 1578735. PMID 16754604.[പ്രവർത്തിക്കാത്ത കണ്ണി]
- ↑ Chappe B, Albrecht P, Michaelis W (1982). "Polar Lipids of Archaebacteria in Sediments and Petroleums". Science. 217 (4554): 65–66. Bibcode:1982Sci...217...65C. doi:10.1126/science.217.4554.65. PMID 17739984.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Brocks JJ, Logan GA, Buick R, Summons RE (1999). "Archean molecular fossils and the early rise of eukaryotes". Science. 285 (5430): 1033–6. doi:10.1126/science.285.5430.1033. PMID 10446042.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Rasmussen B, Fletcher IR, Brocks JJ, Kilburn MR (2008). "Reassessing the first appearance of eukaryotes and cyanobacteria". Nature. 455 (7216): 1101–4. Bibcode:2008Natur.455.1101R. doi:10.1038/nature07381. PMID 18948954.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Hahn, Jürgen (1986). "Traces of Archaebacteria in ancient sediments". System Applied Microbiology. 7 (Archaebacteria '85 Proceedings): 178–83.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help) - ↑ Wang M, Yafremava LS, Caetano-Anollés D, Mittenthal JE, Caetano-Anollés G (2007). "Reductive evolution of architectural repertoires in proteomes and the birth of the tripartite world". Genome Res. 17 (11): 1572–85. doi:10.1101/gr.6454307. PMC 2045140. PMID 17908824.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (2006). "Toward automatic reconstruction of a highly resolved tree of life". Science. 311 (5765): 1283–7. Bibcode:2006Sci...311.1283C. doi:10.1126/science.1123061. PMID 16513982.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Woese CR, Gupta R (1981). "Are archaebacteria merely derived 'prokaryotes'?". Nature. 289 (5793): 95–6. Bibcode:1981Natur.289...95W. doi:10.1038/289095a0. PMID 6161309.
- ↑ Woese C (1998). "The universal ancestor". Proc. Natl. Acad. Sci. U.S.A. 95 (12): 6854–9. Bibcode:1998PNAS...95.6854W. doi:10.1073/pnas.95.12.6854. PMC 22660. PMID 9618502. Archived from the original on 2019-09-18. Retrieved 2012-03-13.
- ↑ Gupta RS (2000). "The natural evolutionary relationships among prokaryotes". Crit. Rev. Microbiol. 26 (2): 111–31. doi:10.1080/10408410091154219. PMID 10890353.
- ↑ C. Michael Hogan. 2010. Virus. Encyclopedia of Earth Archived 2020-01-26 at the Wayback Machine.. Editors: Cutler Cleveland and Sidney Draggan
- ↑ Gribaldo S, Brochier-Armanet C (2006). "The origin and evolution of Archaea: a state of the art". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1470): 1007–22. doi:10.1098/rstb.2006.1841. PMC 1578729. PMID 16754611. Archived from the original on 2012-06-04. Retrieved 2012-03-13.
- ↑ 35.0 35.1 Woese CR (1 March 1994). "There must be a prokaryote somewhere: microbiology's search for itself". Microbiol. Rev. 58 (1): 1–9. PMC 372949. PMID 8177167. Archived from the original on 2019-09-18. Retrieved 2012-03-13.
- ↑ Lake JA (1988). "Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences". Nature. 331 (6152): 184–6. Bibcode:1988Natur.331..184L. doi:10.1038/331184a0. PMID 3340165.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Gouy M, Li WH (1989). "Phylogenetic analysis based on rRNA sequences supports the archaebacterial rather than the eocyte tree". Nature. 339 (6220): 145–7. Bibcode:1989Natur.339..145G. doi:10.1038/339145a0. PMID 2497353.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Yutin N, Makarova KS, Mekhedov SL, Wolf YI, Koonin EV (2008). "The deep archaeal roots of eukaryotes". Mol. Biol. Evol. 25 (8): 1619–30. doi:10.1093/molbev/msn108. PMC 2464739. PMID 18463089. Archived from the original on 2009-05-03. Retrieved 2012-03-13.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Lake JA. (1988). "Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences". Nature. 331 (6152): 184–6. Bibcode:1988Natur.331..184L. doi:10.1038/331184a0. PMID 3340165.
- ↑ 40.0 40.1 40.2 40.3 Krieg, Noel (2005). Bergey's Manual of Systematic Bacteriology. US: Springer. pp. 21–6. ISBN 978-0-387-24143-2.
- ↑ Barns, Sue and Burggraf, Siegfried. (1997) Crenarchaeota Archived 2012-05-02 at the Wayback Machine.. Version 01 January 1997. in The Tree of Life Web Project
- ↑ Walsby, A.E. (1980). "A square bacterium". Nature. 283 (5742): 69–71. Bibcode:1980Natur.283...69W. doi:10.1038/283069a0.
- ↑ Hara F, Yamashiro K, Nemoto N; et al. (2007). "An actin homolog of the archaeon Thermoplasma acidophilum that retains the ancient characteristics of eukaryotic actin". J. Bacteriol. 189 (5): 2039–45. doi:10.1128/JB.01454-06. PMC 1855749. PMID 17189356. Archived from the original on 2020-05-27. Retrieved 2012-03-13.
{{cite journal}}: Explicit use of et al. in:|author=(help)CS1 maint: multiple names: authors list (link) - ↑ Trent JD, Kagawa HK, Yaoi T, Olle E, Zaluzec NJ (1997). "Chaperonin filaments: the archaeal cytoskeleton?". Proc. Natl. Acad. Sci. U.S.A. 94 (10): 5383–8. Bibcode:1997PNAS...94.5383T. doi:10.1073/pnas.94.10.5383. PMC 24687. PMID 9144246. Archived from the original on 2020-05-27. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Hixon WG, Searcy DG (1993). "Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts". BioSystems. 29 (2–3): 151–60. doi:10.1016/0303-2647(93)90091-P. PMID 8374067.
- ↑ 46.0 46.1 Golyshina OV, Pivovarova TA, Karavaiko GI; et al. (1 May 2000). "Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea". Int. J. Syst. Evol. Microbiol. 50 (3): 997–1006. PMID 10843038. Archived from the original on 2023-11-13. Retrieved 2012-03-13.
{{cite journal}}: Explicit use of et al. in:|author=(help)CS1 maint: multiple names: authors list (link) - ↑ Hall-Stoodley L, Costerton JW, Stoodley P (2004). "Bacterial biofilms: from the natural environment to infectious diseases". Nat. Rev. Microbiol. 2 (2): 95–108. doi:10.1038/nrmicro821. PMID 15040259.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Kuwabara T, Minaba M, Iwayama Y; et al. (2005). "Thermococcus coalescens sp. nov., a cell-fusing hyperthermophilic archaeon from Suiyo Seamount". Int. J. Syst. Evol. Microbiol. 55 (Pt 6): 2507–14. doi:10.1099/ijs.0.63432-0. PMID 16280518.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Nickell S, Hegerl R, Baumeister W, Rachel R (2003). "Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography". J. Struct. Biol. 141 (1): 34–42. doi:10.1016/S1047-8477(02)00581-6. PMID 12576018. Archived from the original on 2018-06-27. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Horn C, Paulmann B, Kerlen G, Junker N, Huber H (15 August 1999). "In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope". J. Bacteriol. 181 (16): 5114–8. PMC 94007. PMID 10438790. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Rudolph C, Wanner G, Huber R (2001). "Natural communities of novel archaea and bacteria growing in cold sulfurous springs with a string-of-pearls-like morphology". Appl. Environ. Microbiol. 67 (5): 2336–44. doi:10.1128/AEM.67.5.2336-2344.2001. PMC 92875. PMID 11319120.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ 52.0 52.1 Thomas NA, Bardy SL, Jarrell KF (2001). "The archaeal flagellum: a different kind of prokaryotic motility structure". FEMS Microbiol. Rev. 25 (2): 147–74. doi:10.1111/j.1574-6976.2001.tb00575.x. PMID 11250034.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Rachel R, Wyschkony I, Riehl S, Huber H (2002). "The ultrastructure of Ignicoccus: evidence for a novel outer membrane and for intracellular vesicle budding in an archaeon" (PDF). Archaea. 1 (1): 9–18. doi:10.1155/2002/307480. PMC 2685547. PMID 15803654. Archived (PDF) from the original on 2009-02-24. Retrieved 2012-03-13.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) CS1 maint: unflagged free DOI (link) - ↑ 54.0 54.1 Koga Y, Morii H (2007). "Biosynthesis of ether-type polar lipids in archaea and evolutionary considerations". Microbiol. Mol. Biol. Rev. 71 (1): 97–120. doi:10.1128/MMBR.00033-06. PMC 1847378. PMID 17347520. Archived from the original on 2020-05-27. Retrieved 2012-03-13.
- ↑ De Rosa M, Gambacorta A, Gliozzi A (1 March 1986). "Structure, biosynthesis, and physicochemical properties of archaebacterial lipids". Microbiol. Rev. 50 (1): 70–80. PMC 373054. PMID 3083222. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Albers SV, van de Vossenberg JL, Driessen AJ, Konings WN (2000). "Adaptations of the archaeal cell membrane to heat stress". Front. Biosci. 5: D813–20. doi:10.2741/albers. PMID 10966867. Archived from the original on 2012-10-27. Retrieved 2012-03-13.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Damsté JS, Schouten S, Hopmans EC, van Duin AC, Geenevasen JA (2002). "Crenarchaeol: the characteristic core glycerol dibiphytanyl glycerol tetraether membrane lipid of cosmopolitan pelagic crenarchaeota". J. Lipid Res. 43 (10): 1641–51. doi:10.1194/jlr.M200148-JLR200. PMID 12364548. Archived from the original on 2020-05-27. Retrieved 2012-03-13.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Koga Y, Morii H (2005). "Recent advances in structural research on ether lipids from archaea including comparative and physiological aspects". Biosci. Biotechnol. Biochem. 69 (11): 2019–34. doi:10.1271/bbb.69.2019. PMID 16306681. Archived from the original on 2008-12-31. Retrieved 2012-03-13.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Hanford MJ, Peeples TL (2002). "Archaeal tetraether lipids: unique structures and applications". Appl. Biochem. Biotechnol. 97 (1): 45–62. doi:10.1385/ABAB:97:1:45. PMID 11900115.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Macalady JL, Vestling MM, Baumler D, Boekelheide N, Kaspar CW, Banfield JF (2004). "Tetraether-linked membrane monolayers in Ferroplasma spp: a key to survival in acid". Extremophiles. 8 (5): 411–9. doi:10.1007/s00792-004-0404-5. PMID 15258835.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Sára M, Sleytr UB (2000). "S-Layer proteins". J. Bacteriol. 182 (4): 859–68. doi:10.1128/JB.182.4.859-868.2000. PMC 94357. PMID 10648507. Archived from the original on 2020-05-27. Retrieved 2012-03-13.
- ↑ Engelhardt H, Peters J (1998). "Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions". J Struct Biol. 124 (2–3): 276–302. doi:10.1006/jsbi.1998.4070. PMID 10049812.
- ↑ 63.0 63.1 Kandler, O; König, H (1998). "Cell wall polymers in Archaea (Archaebacteria)" (PDF). Cellular and Molecular Life Sciences (CMLS). 54 (4): 305–308. doi:10.1007/s000180050156.[പ്രവർത്തിക്കാത്ത കണ്ണി]
- ↑ Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. p. 32. ISBN 0-19-511183-4.
- ↑ Gophna U, Ron EZ, Graur D (2003). "Bacterial type III secretion systems are ancient and evolved by multiple horizontal-transfer events". Gene. 312: 151–63. doi:10.1016/S0378-1119(03)00612-7. PMID 12909351. Archived from the original on 2017-11-22. Retrieved 2012-03-13.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Nguyen L, Paulsen IT, Tchieu J, Hueck CJ, Saier MH (2000). "Phylogenetic analyses of the constituents of Type III protein secretion systems". J. Mol. Microbiol. Biotechnol. 2 (2): 125–44. PMID 10939240.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Ng SY, Chaban B, Jarrell KF (2006). "Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications". J. Mol. Microbiol. Biotechnol. 11 (3–5): 167–91. doi:10.1159/000094053. PMID 16983194.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Bardy SL, Ng SY, Jarrell KF (2003). "Prokaryotic motility structures". Microbiology (Reading, Engl.). 149 (Pt 2): 295–304. doi:10.1099/mic.0.25948-0. PMID 12624192.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ 69.0 69.1 69.2 Valentine DL (2007). "Adaptations to energy stress dictate the ecology and evolution of the Archaea". Nat. Rev. Microbiol. 5 (4): 316–23. doi:10.1038/nrmicro1619. PMID 17334387.
- ↑ 70.0 70.1 70.2 Schäfer G, Engelhard M, Müller V (1 September 1999). "Bioenergetics of the Archaea". Microbiol. Mol. Biol. Rev. 63 (3): 570–620. PMC 103747. PMID 10477309. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Zillig W (1991). "Comparative biochemistry of Archaea and Bacteria". Curr. Opin. Genet. Dev. 1 (4): 544–51. doi:10.1016/S0959-437X(05)80206-0. PMID 1822288.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Romano A, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Res Microbiol. 147 (6–7): 448–55. doi:10.1016/0923-2508(96)83998-2. PMID 9084754.
- ↑ Based on PDB 1FBB Archived 2016-03-03 at the Wayback Machine.. Data published in Subramaniam S, Henderson R (2000). "Molecular mechanism of vectorial proton translocation by bacteriorhodopsin". Nature. 406 (6796): 653–7. doi:10.1038/35020614. PMID 10949309.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Koch A (1998). "How did bacteria come to be?". Adv Microb Physiol. 40: 353–99. doi:10.1016/S0065-2911(08)60135-6. PMID 9889982.
- ↑ DiMarco AA, Bobik TA, Wolfe RS (1990). "Unusual coenzymes of methanogenesis". Annu. Rev. Biochem. 59: 355–94. doi:10.1146/annurev.bi.59.070190.002035. PMID 2115763.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Klocke M, Nettmann E, Bergmann I; et al. (2008). "Characterization of the methanogenic Archaea within two-phase biogas reactor systems operated with plant biomass". Syst. Appl. Microbiol. 31 (3): 190–205. doi:10.1016/j.syapm.2008.02.003. PMID 18501543.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Mueller-Cajar O, Badger MR (2007). "New roads lead to Rubisco in archaebacteria". Bioessays. 29 (8): 722–4. doi:10.1002/bies.20616. PMID 17621634.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Berg IA, Kockelkorn D, Buckel W, Fuchs G (2007). "A 3-hydroxypropionate/4-hydroxybutyrate autotrophic carbon dioxide assimilation pathway in Archaea". Science. 318 (5857): 1782–6. Bibcode:2007Sci...318.1782B. doi:10.1126/science.1149976. PMID 18079405.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Thauer RK (2007). "Microbiology. A fifth pathway of carbon fixation". Science. 318 (5857): 1732–3. doi:10.1126/science.1152209. PMID 18079388.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Bryant DA, Frigaard NU (2006). "Prokaryotic photosynthesis and phototrophy illuminated". Trends Microbiol. 14 (11): 488–96. doi:10.1016/j.tim.2006.09.001. PMID 16997562.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Könneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA (2005). "Isolation of an autotrophic ammonia-oxidizing marine archaeon". Nature. 437 (7058): 543–6. Bibcode:2005Natur.437..543K. doi:10.1038/nature03911. PMID 16177789.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Francis CA, Beman JM, Kuypers MM (2007). "New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation". ISME J. 1 (1): 19–27. doi:10.1038/ismej.2007.8. PMID 18043610.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Lanyi JK (2004). "Bacteriorhodopsin". Annu. Rev. Physiol. 66: 665–88. doi:10.1146/annurev.physiol.66.032102.150049. PMID 14977418.
- ↑ 84.0 84.1 Allers T, Mevarech M (2005). "Archaeal genetics - the third way". Nat. Rev. Genet. 6 (1): 58–73. doi:10.1038/nrg1504. PMID 15630422.
- ↑ Galagan JE, Nusbaum C, Roy A; et al. (2002). "The genome of M. acetivorans reveals extensive metabolic and physiological diversity". Genome Res. 12 (4): 532–42. doi:10.1101/gr.223902. PMC 187521. PMID 11932238.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Waters E; et al. (2003). "The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism". Proc. Natl. Acad. Sci. U.S.A. 100 (22): 12984–8. Bibcode:2003PNAS..10012984W. doi:10.1073/pnas.1735403100. PMC 240731. PMID 14566062. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: Explicit use of et al. in:|author=(help) - ↑ Schleper C, Holz I, Janekovic D, Murphy J, Zillig W (1 August 1995). "A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating". J. Bacteriol. 177 (15): 4417–26. PMC 177192. PMID 7635827. Archived from the original on 2012-05-29. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Sota M; Top EM (2008). "Horizontal Gene Transfer Mediated by Plasmids". Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
{{cite book}}:|access-date=requires|url=(help);|archive-url=requires|url=(help); External link in|chapterurl=|chapterurl=ignored (|chapter-url=suggested) (help)CS1 maint: multiple names: authors list (link) - ↑ Xiang X, Chen L, Huang X, Luo Y, She Q, Huang L (2005). "Sulfolobus tengchongensis spindle-shaped virus STSV1: virus-host interactions and genomic features". J. Virol. 79 (14): 8677–86. doi:10.1128/JVI.79.14.8677-8686.2005. PMC 1168784. PMID 15994761. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Prangishvili D, Forterre P, Garrett RA (2006). "Viruses of the Archaea: a unifying view". Nat. Rev. Microbiol. 4 (11): 837–48. doi:10.1038/nrmicro1527. PMID 17041631.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Prangishvili D, Garrett RA (2004). "Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses". Biochem. Soc. Trans. 32 (Pt 2): 204–8. doi:10.1042/BST0320204. PMID 15046572. Archived from the original on 2006-04-18. Retrieved 2012-03-13.
- ↑ Pietilä MK, Roine E, Paulin L, Kalkkinen N, Bamford DH (2009). "An ssDNA virus infecting archaea; A new lineage of viruses with a membrane envelope". Mol. Microbiol. 72 (2): 307–19. doi:10.1111/j.1365-2958.2009.06642.x. PMID 19298373.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (2005). "Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements". J. Mol. Evol. 60 (2): 174–82. doi:10.1007/s00239-004-0046-3. PMID 15791728.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (2006). "A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action". Biol. Direct. 1: 7. doi:10.1186/1745-6150-1-7. PMC 1462988. PMID 16545108.
{{cite journal}}: CS1 maint: multiple names: authors list (link) CS1 maint: unflagged free DOI (link) - ↑ Graham DE, Overbeek R, Olsen GJ, Woese CR (2000). "An archaeal genomic signature". Proc. Natl. Acad. Sci. U.S.A. 97 (7): 3304–8. Bibcode:2000PNAS...97.3304G. doi:10.1073/pnas.050564797. PMC 16234. PMID 10716711.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ 96.0 96.1 Gaasterland T (1999). "Archaeal genomics". Curr. Opin. Microbiol. 2 (5): 542–7. doi:10.1016/S1369-5274(99)00014-4. PMID 10508726.
- ↑ Werner F (2007). "Structure and function of archaeal RNA polymerases". Mol. Microbiol. 65 (6): 1395–404. doi:10.1111/j.1365-2958.2007.05876.x. PMID 17697097.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Aravind L, Koonin EV (1999). "DNA-binding proteins and evolution of transcription regulation in the archaea". Nucleic Acids Res. 27 (23): 4658–70. doi:10.1093/nar/27.23.4658. PMC 148756. PMID 10556324. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
- ↑ Lykke-Andersen J, Aagaard C, Semionenkov M, Garrett RA (1997). "Archaeal introns: splicing, intercellular mobility and evolution". Trends Biochem. Sci. 22 (9): 326–31. doi:10.1016/S0968-0004(97)01113-4. PMID 9301331.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Watanabe Y, Yokobori S, Inaba T; et al. (2002). "Introns in protein-coding genes in Archaea". FEBS Lett. 510 (1–2): 27–30. doi:10.1016/S0014-5793(01)03219-7. PMID 11755525.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Yoshinari S, Itoh T, Hallam SJ; et al. (2006). "Archaeal pre-mRNA splicing: a connection to hetero-oligomeric splicing endonuclease". Biochem. Biophys. Res. Commun. 346 (3): 1024–32. doi:10.1016/j.bbrc.2006.06.011. PMID 16781672.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ 102.0 102.1 Bernander R (1998). "Archaea and the cell cycle". Mol. Microbiol. 29 (4): 955–61. doi:10.1046/j.1365-2958.1998.00956.x. PMID 9767564.
- ↑ Kelman LM, Kelman Z (2004). "Multiple origins of replication in archaea". Trends Microbiol. 12 (9): 399–401. doi:10.1016/j.tim.2004.07.001. PMID 15337158.
- ↑ Onyenwoke RU, Brill JA, Farahi K, Wiegel J (2004). "Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch ( Firmicutes)". Arch. Microbiol. 182 (2–3): 182–92. doi:10.1007/s00203-004-0696-y. PMID 15340788.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Kostrikina NA, Zvyagintseva IS, Duda VI. (1991). "Cytological peculiarities of some extremely halophilic soil archaeobacteria". Arch. Microbiol. 156 (5): 344–49. doi:10.1007/BF00248708.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ DeLong EF, Pace NR (2001). "Environmental diversity of bacteria and archaea". Syst. Biol. 50 (4): 470–8. doi:10.1080/106351501750435040. PMID 12116647.
- ↑ 107.0 107.1 Pikuta EV, Hoover RB, Tang J (2007). "Microbial extremophiles at the limits of life". Crit. Rev. Microbiol. 33 (3): 183–209. doi:10.1080/10408410701451948. PMID 17653987.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Madigan MT, Martino JM (2006). Brock Biology of Microorganisms (11th ed.). Pearson. p. 136. ISBN 0-13-196893-9.
- ↑ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (2008). "Cell proliferation at 122 °C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation". Proc Natl Acad Sci USA. 105 (31): 10949–54. Bibcode:2008PNAS..10510949T. doi:10.1073/pnas.0712334105. PMC 2490668. PMID 18664583.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Ciaramella M, Napoli A, Rossi M (2005). "Another extreme genome: how to live at pH 0". Trends Microbiol. 13 (2): 49–51. doi:10.1016/j.tim.2004.12.001. PMID 15680761.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Javaux EJ (2006). "Extreme life on Earth—past, present and possibly beyond". Res. Microbiol. 157 (1): 37–48. doi:10.1016/j.resmic.2005.07.008. PMID 16376523.
- ↑ Nealson KH (1999). "Post-Viking microbiology: new approaches, new data, new insights" (PDF). Orig Life Evol Biosph. 29 (1): 73–93. doi:10.1023/A:1006515817767. PMID 11536899. Archived from the original (PDF) on 2019-10-16. Retrieved 2012-06-12.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Davies PC (1996). "The transfer of viable microorganisms between planets". Ciba Found. Symp. 202: 304–14, discussion 314–7. PMID 9243022.
- ↑ López-García P, López-López A, Moreira D, Rodríguez-Valera F (2001). "Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front". FEMS Microbiol. Ecol. 36 (2–3): 193–202. PMID 11451524.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Karner MB, DeLong EF, Karl DM (2001). "Archaeal dominance in the mesopelagic zone of the Pacific Ocean". Nature. 409 (6819): 507–10. doi:10.1038/35054051. PMID 11206545.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Giovannoni SJ, Stingl U. (2005). "Molecular diversity and ecology of microbial plankton". Nature. 427 (7057): 343–8. Bibcode:2005Natur.437..343G. doi:10.1038/nature04158. PMID 16163344.
- ↑ DeLong EF, Karl DM (2005). "Genomic perspectives in microbial oceanography". Nature. 437 (7057): 336–42. Bibcode:2005Natur.437..336D. doi:10.1038/nature04157. PMID 16163343.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Konneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA. (2005). "Isolation of an autotrophic ammonia-oxidizing marine archaeon". Nature. 437 (7057): 543–6. Bibcode:2005Natur.437..543K. doi:10.1038/nature03911. PMID 16177789.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Agogué, H; Brink, M; Dinasquet, J; Herndl, GJ (2008). "Major gradients in putatively nitrifying and non-nitrifying Archaea in the deep North Atlantic". Nature. 456 (7223): 788–791. Bibcode:2008Natur.456..788A. doi:10.1038/nature07535. PMID 19037244.
{{cite journal}}: More than one of|author=and|last1=specified (help) - ↑ Teske A, Sørensen KB (2008). "Uncultured archaea in deep marine subsurface sediments: have we caught them all?". ISME J. 2 (1): 3–18. doi:10.1038/ismej.2007.90. PMID 18180743.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Lipp JS, Morono Y, Inagaki F, Hinrichs KU (2008). "Significant contribution of Archaea to extant biomass in marine subsurface sediments". Nature. 454 (7207): 991–4. Bibcode:2008Natur.454..991L. doi:10.1038/nature07174. PMID 18641632.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Cabello P, Roldán MD, Moreno-Vivián C (2004). "Nitrate reduction and the nitrogen cycle in archaea". Microbiology (Reading, Engl.). 150 (Pt 11): 3527–46. doi:10.1099/mic.0.27303-0. PMID 15528644. Archived from the original on 2011-06-07. Retrieved 2012-06-12.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Mehta MP, Baross JA (2006). "Nitrogen fixation at 92 degrees C by a hydrothermal vent archaeon". Science. 314 (5806): 1783–6. Bibcode:2006Sci...314.1783M. doi:10.1126/science.1134772. PMID 17170307.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Francis CA, Beman JM, Kuypers MM (2007). "New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation". ISME J. 1 (1): 19–27. doi:10.1038/ismej.2007.8. PMID 18043610.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Coolen MJ, Abbas B, van Bleijswijk J; et al. (2007). "Putative ammonia-oxidizing Crenarchaeota in suboxic waters of the Black Sea: a basin-wide ecological study using 16S ribosomal and functional genes and membrane lipids". Environ. Microbiol. 9 (4): 1001–16. doi:10.1111/j.1462-2920.2006.01227.x. PMID 17359272.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Leininger S, Urich T, Schloter M; et al. (2006). "Archaea predominate among ammonia-oxidizing prokaryotes in soils". Nature. 442 (7104): 806–9. Bibcode:2006Natur.442..806L. doi:10.1038/nature04983. PMID 16915287.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Baker, B. J; Banfield, J. F (2003). "Microbial communities in acid mine drainage". FEMS Microbiology Ecology. 44 (2): 139–152. doi:10.1016/S0168-6496(03)00028-X. PMID 19719632. Archived from the original on 2023-11-13. Retrieved 2019-10-27.
- ↑ Schimel J (2004). "Playing scales in the methane cycle: from microbial ecology to the globe". Proc. Natl. Acad. Sci. U.S.A. 101 (34): 12400–1. Bibcode:2004PNAS..10112400S. doi:10.1073/pnas.0405075101. PMC 515073. PMID 15314221. Archived from the original on 2019-10-16. Retrieved 2012-06-12.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ "EDGAR 3.2 Fast Track 2000". Archived from the original on 2008-05-21. Retrieved 2008-06-26.
- ↑ "Annual Greenhouse Gas Index (AGGI) Indicates Sharp Rise in Carbon Dioxide and Methane in 2007". 2008-04-23. Archived from the original on 2008-05-14. Retrieved 2008-06-26.
- ↑ "Trace Gases: Current Observations, Trends, and Budgets". Climate Change 2001. United Nations Environment Programme. Archived from the original on 2012-07-28. Retrieved 2012-06-12.
- ↑ Eckburg P, Lepp P, Relman D (2003). "Archaea and their potential role in human disease". Infect Immun. 71 (2): 591–6. doi:10.1128/IAI.71.2.591-596.2003. PMC 145348. PMID 12540534.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Cavicchioli R, Curmi P, Saunders N, Thomas T (2003). "Pathogenic archaea: do they exist?". Bioessays. 25 (11): 1119–28. doi:10.1002/bies.10354. PMID 14579252.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Lepp P, Brinig M, Ouverney C, Palm K, Armitage G, Relman D (2004). "Methanogenic Archaea and human periodontal disease". Proc Natl Acad Sci USA. 101 (16): 6176–81. Bibcode:2004PNAS..101.6176L. doi:10.1073/pnas.0308766101. PMC 395942. PMID 15067114.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Vianna ME, Conrads G, Gomes BP, Horz HP (2006). "Identification and quantification of archaea involved in primary endodontic infections". J. Clin. Microbiol. 44 (4): 1274–82. doi:10.1128/JCM.44.4.1274-1282.2006. PMC 1448633. PMID 16597851. Archived from the original on 2019-10-16. Retrieved 2012-06-12.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Waters E, Hohn MJ, Ahel I; et al. (2003). "The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism". Proc. Natl. Acad. Sci. U.S.A. 100 (22): 12984–8. Bibcode:2003PNAS..10012984W. doi:10.1073/pnas.1735403100. PMC 240731. PMID 14566062. Archived from the original on 2019-10-16. Retrieved 2012-03-13.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Jahn U, Gallenberger M, Paper W; et al. (2008). "Nanoarchaeum equitans and Ignicoccus hospitalis: new insights into a unique, intimate association of two archaea". J. Bacteriol. 190 (5): 1743–50. doi:10.1128/JB.01731-07. PMC 2258681. PMID 18165302. Archived from the original on 2020-05-27. Retrieved 2012-06-12.
{{cite journal}}: Explicit use of et al. in:|author=(help); Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Baker BJ, Comolli LR, Dick GJ, Hauser LJ, Hyatt D, Dill BD, Land ML, VerBerkmoes NC, Hettich RL, Banfield JF (2010). "Enigmatic, ultrasmall, uncultivated Archaeaa". Proc. Natl. Acad. Sci. U.S.A. 107 (19): 8806–8811. doi:10.1073/pnas.0914470107. PMC 2889320. PMID 20421484. Archived from the original on 2021-02-10. Retrieved 2012-06-12.
{{cite journal}}: Unknown parameter|month=ignored (help)CS1 maint: multiple names: authors list (link) - ↑ Breithaupt H (2001). "The hunt for living gold. The search for organisms in extreme environments yields useful enzymes for industry". EMBO Rep. 2 (11): 968–71. doi:10.1093/embo-reports/kve238. PMC 1084137. PMID 11713183.
- ↑ 140.0 140.1 Egorova K, Antranikian G (2005). "Industrial relevance of thermophilic Archaea". Curr. Opin. Microbiol. 8 (6): 649–55. doi:10.1016/j.mib.2005.10.015. PMID 16257257.
- ↑ Synowiecki J, Grzybowska B, Zdziebło A (2006). "Sources, properties and suitability of new thermostable enzymes in food processing". Crit Rev Food Sci Nutr. 46 (3): 197–205. doi:10.1080/10408690590957296. PMID 16527752.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Jenney FE, Adams MW (2008). "The impact of extremophiles on structural genomics (and vice versa)". Extremophiles. 12 (1): 39–50. doi:10.1007/s00792-007-0087-9. PMID 17563834.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Schiraldi C, Giuliano M, De Rosa M (2002). "Perspectives on biotechnological applications of archaea" (PDF). Archaea. 1 (2): 75–86. doi:10.1155/2002/436561. PMC 2685559. PMID 15803645. Archived from the original (PDF) on 2013-08-26. Retrieved 2012-06-15.
{{cite journal}}: CS1 maint: multiple names: authors list (link) CS1 maint: unflagged free DOI (link) - ↑ Norris PR, Burton NP, Foulis NA (2000). "Acidophiles in bioreactor mineral processing". Extremophiles. 4 (2): 71–6. doi:10.1007/s007920050139. PMID 10805560.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ O'Connor EM, Shand RF (2002). "Halocins and sulfolobicins: the emerging story of archaeal protein and peptide antibiotics". J. Ind. Microbiol. Biotechnol. 28 (1): 23–31. doi:10.1038/sj/jim/7000190. PMID 11938468.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Shand RF; Leyva KJ (2008). "Archaeal Antimicrobials: An Undiscovered Country". In Blum P (ed.) (ed.). Archaea: New Models for Prokaryotic Biology. Caister Academic Press. ISBN 978-1-904455-27-1.
{{cite book}}:|editor=has generic name (help)CS1 maint: multiple names: authors list (link)
കൂടുതൽ വായനക്ക്[തിരുത്തുക]
- Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. ISBN 0-19-511183-4.
- Martinko JM, Madigan MT (2005). Brock Biology of Microorganisms (11th ed.). Englewood Cliffs, N.J: Prentice Hall. ISBN 0-13-144329-1.
- Garrett RA, Klenk H (2005). Archaea: Evolution, Physiology and Molecular Biology. WileyBlackwell. ISBN 1-4051-4404-1.
- Cavicchioli R (2007). Archaea: Molecular and Cellular Biology. American Society for Microbiology. ISBN 1-55581-391-7.
- Blum P (editor) (2008). Archaea: New Models for Prokaryotic Biology. Caister Academic Press. ISBN 978-1-904455-27-1.
{{cite book}}:|author=has generic name (help) - Lipps G (2008). "Archaeal Plasmids". Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
- Sapp, Jan (2009). The New Foundations of Evolution. On the Tree of Life. New York: Oxford University Press. ISBN 0-19-538850-X.
- Schaechter, M (2009). Archaea (Overview) in The Desk Encyclopedia of Microbiology, 2nd edition. San Diego and London: Elsevier Academic Press. ISBN 978-0-12-374980-2.
പുറത്തേക്കുള്ള കണ്ണികൾ[തിരുത്തുക]
പൊതുവായവ
- Introduction to the Archaea, ecology, systematics and morphology
- Oceans of Archaea[പ്രവർത്തിക്കാത്ത കണ്ണി] – E.F. DeLong, ASM News, 2003
വർഗ്ഗീകരണം
- NCBI taxonomy page on Archaea
- Genera of the domain Archaea – list of Prokaryotic names with Standing in Nomenclature
- Tree of Life illustration showing how Archaea relates to other lifeforms Archived 2015-02-10 at the Wayback Machine.
- Shotgun sequencing finds nanoorganisms – discovery of the ARMAN group of archaea
ജനിതകം
